收藏:表观遗传学常用工具及数据库
2020-06-13 李伟、彭宇中、朱兴星、匡正 表观遗传学习小组
生物信息学在表观遗传领域里面的应用,还是依据表观遗传学的分类的,包括 DNA 甲基化修饰、组蛋白修饰,还有非编码 RNA 的调控,在我的 p 里面呢,我会从下面的三个部分来帮助大家走进生物信息学。
生物信息学在表观遗传领域里面的应用,还是依据表观遗传学的分类的,包括 DNA 甲基化修饰、组蛋白修饰,还有非编码 RNA 的调控,在我的 p 里面呢,我会从下面的三个部分来帮助大家走进生物信息学。
第一部分:ENCODE 及 Roadmap 数据库的介绍。我们做表观遗传的时候可能都会涉及到这两大计划,这两大计划耗费了很多钱和人力来完成。我们怎样能更好地使用和浏览这两大计划里面的一些数据和数据库。我会提供 UCSC 王艇老师(圣路易斯华盛顿大学教授)组开发的一个 server 工具,帮助大家合理地使用和浏览这两大计划产生的数据。其实很多人认为这两大计划产生的数据就足够的。
第二部分:RNA-seq 数据获取及分析流程。RNA-seq 我会给大家大概讲一下,怎么从网站上下载一些别人已经测过的或者 paper 已经发表的 RNA-seq 数据。当我拿到了别人的数据以后,我应该用哪些流程来评估和分析这些数据,因为表达数据我们大部分研究都是要用到的;还有就是 RNA-seq 里面现在有一些主流的分析套路,包括 TopHat、TopHat2 等。2016 年 5 月还出了一个新的方法,文章里评估它的性能不输于 TopHat2,我会把它的 map 原理分享给大家。
最后一部分:表观遗传学常用数据库介绍。这部分我展示的时间并不长,我会依据表观遗传学不同的分类,每个部分推荐大家几个常用的数据库,包括里面也有我自己写的 server。看的时候,我通常会把这些网站归为两类:一类是“淘宝”类的网页,一类是需要一定的编程基础的。所以第三个部分,我推荐给大家的基本都是“淘宝”类的网页,点按式的,界面都很友好的。大家记住都有哪些资源,就 OK 了。
PART 1: UCSC 及 Roadmap 数据库的介绍
UCSC 数据库:一个存储 ENCODE 计划数据的数据库。如果你做 seq data,不管你是做实验的还是做信息学分析的,你都会用到这个 UCSC 数据库。这里面我会展示几个常用的板块。
UCSC 简介:
1. 给浏览基因组数据提供了可靠和迅速的方式。
2. 数据来源:约有一半的注释信息是 UCSC 通过来自公开的序列数据计算得出,另外一半来自世界各地的科学工作者。
3. 本身并不下任何结论,只是收集各种相关信息供用户参考。
4. 支持数据库检索和序列相似性搜索。
UCSC 网页上面有各种工具,我会给大家简要地介绍一下其中的五个。
Genome Browser
图 1. Genome Browser 首页
我们不管是做计算的,还是做实验的人,都会用到的 Genome 这个 p(或者可点击 Our tools 中的 Genome Browser)。这个 p 你点击进去以后,有很多的选项,都是点按式的。然后我把每个可拉选的菜单都放在这儿了。现在肯定有更新的 version 了,所以我们通常使用 UCSC 数据库的时候,你一定要选好物种,选 version,看它是哪个版本的参考基因组。我们组就曾经出现过情况,我分析的时候是 hg19 的参考基因组及相应的基因位置注释,我们接受结果的同学可能看到的就是 hg38 版本的,他就说这个位置怎么对不上,是不是弄错了。我说应该不会有错,我问他是怎么选的,最后发现他就是因为版本搞错了。大家用的时候一定记住前提,物种、version 一定不能错,要跟你分析的那个参考基因组的版本一定要一致,否则位置会串的。
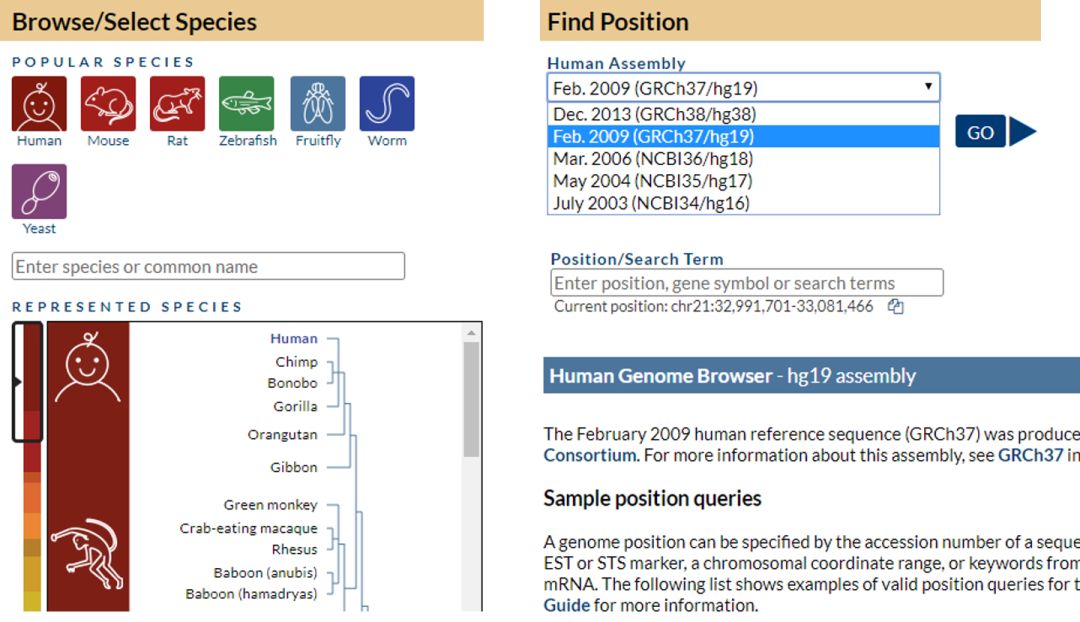
图 2. Genome 板块的界面
这个页面的每个部分,都很容易看懂。在 Position/Search Term 这可以输入一些基因名字,比如说我随便输入一个 BRCA1,它是乳腺癌里面很有名的一个基因,它立刻出现不同的 isoforms,还有一些 non-coding 的 isoform 形式。你可以根据关注的基因组位置,来 filter 这些结果。
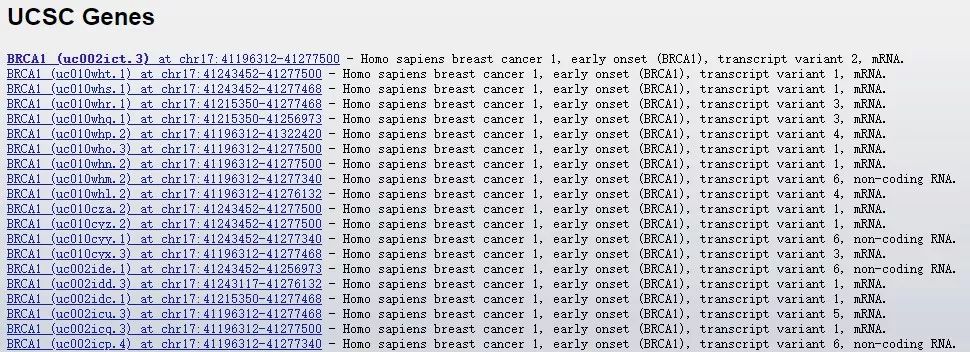
图 3. BRCA1 基因对应的多种 isoforms
如果你确定了其中的一个转录本(下面以 BRCA1 (uc002ict.3) 为例),点开,整个的网页很长,里面包含的信息量很大。上面是一个可视化的部分,下面是一些 tracks ,帮助我们在可视化的部分添加不同的 track。
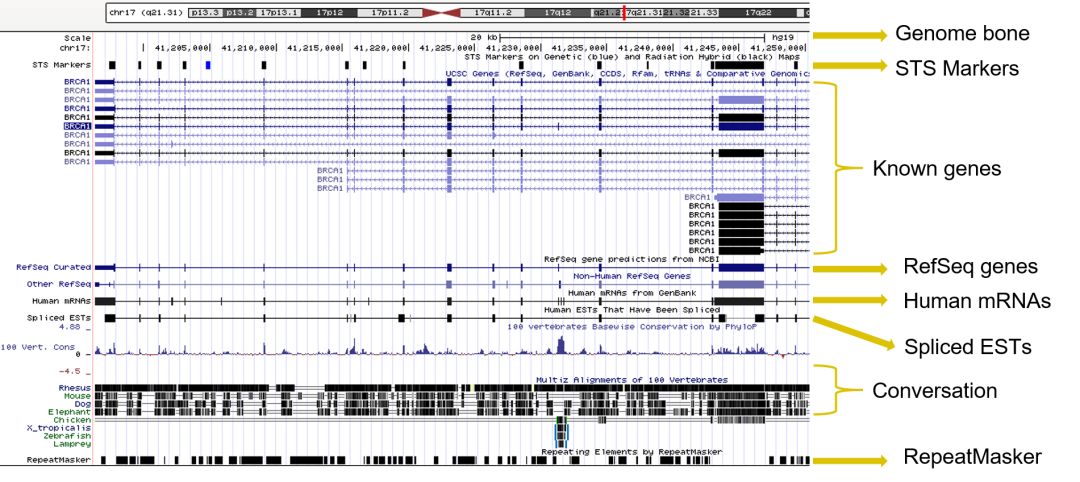
图 4. BRCA1 基因在 Genome Broswer 中的可视化
上面的可视化部分,行包括基因组的 backbone、STS (Sequence-tagged site)marker、不同的 isoform,还有参考基因组。我选的是人类的 mRNA,还有一些同源物种的,因为有的人是做进化的,需要做不同的同源物种,还有 ESTs、保守性(conservation),保守性我等会有一张专门 slide 告诉你这里面不同的颜色、不同的框、不同的形状代表的是什么。

图 5. 基因不同区域和颜色代表不同含义
一个基因有不同的 isoform,不同的 isoform 上有不同的颜色。每一个框就是每一个 exon。最左边的这种有点像“T字形”的都是 UTR 的部分,上面箭头的方向就是它的转录方向。不同的外显子部分的颜色,一共有三种,黑色、深蓝色、浅蓝色(本示例中没有)。黑色表示这一段的表达在 PDB(Protein Data Bank)数据库里面结构已经解析的很清晰了。如果是深蓝色的,说明它大部分的结构在 PDB 里没有很好的解释,但有很多的 paper 含有相关的注释,也就是说它的结构有可能是不知道的。还有一种浅蓝色,意思是它的报道不多,结构也不知道。
中间的部分很好理解,就是一些参考基因组,后面就是保守性。其他部分学起来都很容易,但保守性这部分相对难以理解,原因就是这里面有不同的颜色和形状。
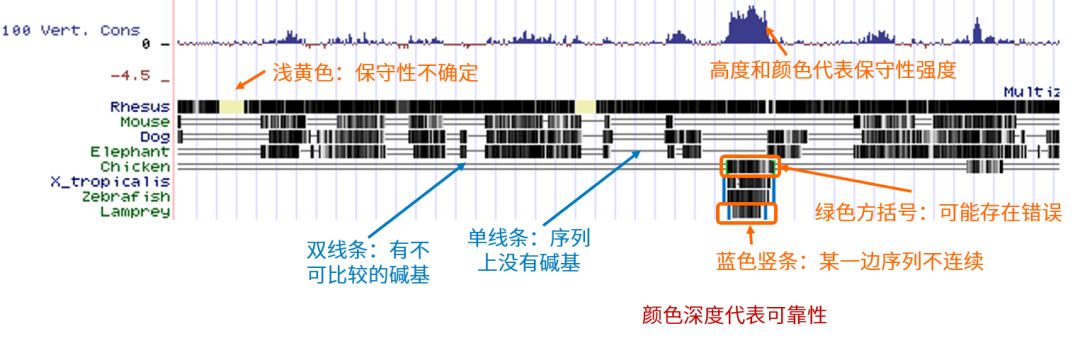
图 6. 序列保守性中不同颜色和形状的具体含义
上图中左侧显示的是不同的物种,图中单线条表示上面是没有序列(碱基)的。如果某一段 mark 上了浅黄色,说明它的保守性方面还没有报道,数据库里面没有备注。保守性的高度和颜色深度,代表它保守性的强度。它的高度越高颜色越深,它的保守性越好,上面有一个整体的评估。比如说我就看其中我关注的这一段它的保守性是不是很好,如果你不是指定物种的,在多个物种里你想看的话,看这个高度和颜色,就能判断保守性是不是很好。还有就是一段被两个蓝色的线条包裹,说明它至少有一边保守性我们是不知道的。如果是被绿色的方括号标注,提示这里面可能是有错误的,是一个有争议的区域。还有一些双线条的,表示有不可比较的碱基。
作为一个可视化的工具,Genome Browser 有一百多个可视化的 tracks,现在应该不止一百多个。我们怎样来筛选和设置这些 tracks 呢?它的设置模式其实就这么几种(dense、hide、squish、pack、full)。我们把这几种显示方式熟悉了,就知道怎么使用了。
Hide:不显示。
Dense:所有信息都在一条直线里显示出来。如果你快速的浏览多个基因的话,或者你要是筛选很多的 tracks 同时看的时候,这是一种相对节约空间的模式。
Full:如果你想选 full 这种模式,它每一种 isoform 就是一行,这样它一个基因就会占用你页面里很长的长度。除非你想深入研究某个关注的基因,才选择这种方式。
Pack:每一项都单独显示,如果有一些项目比较短,后面就会接上其他的,这种方式可以尽量合理地填充整个页面的空白。
Squish:这种模式和 pack 类似,但是是一种压缩模式,高度只有整个 pack 模式的一半,不能看得很细。
我们知道了每一项怎么用,比如说我确定关注 BRCA1 的某一种 isoform,我点击进去里面信息仍然是很多的。所以每次在我讲 UCSC 数据库的时候,我都会千叮咛万嘱咐,你不要在注释页面里到处点,最后你可能会离 UCSC 数据库很远了,因为它会关联到很多个它支持的 server 上面。

图 7. 特定 isoform 的页面中依然蕴含海量信息
一个基因如果你从头到尾,每个都看的很细,我估计要 10min 左右的。在每一个条目里头,它都有一些外部 server 的链接。除非你必须要去看,否则的话你先把整个页面里的信息读好,需要的话再转过去,否则的话会一直在走岔路,离这个网页越来越远。
在 UCSC 里面,通常我们自己会有一些特殊处理的组织或是自己构建的特殊处理的样本的测序数据。这样我分析完以后,我的背景数据库里面没有,我怎么看?比如说我的数据都 map 完了,我现在有一种特殊处理的样本,处理前后的样本做了 RNA-seq,并且做了差异表达分析。做完差异表达以后,我怎么在这个上面看?这就是在 Genome Browser 里面有一个 add custom tracks,这个地方可以上传你自己的数据。
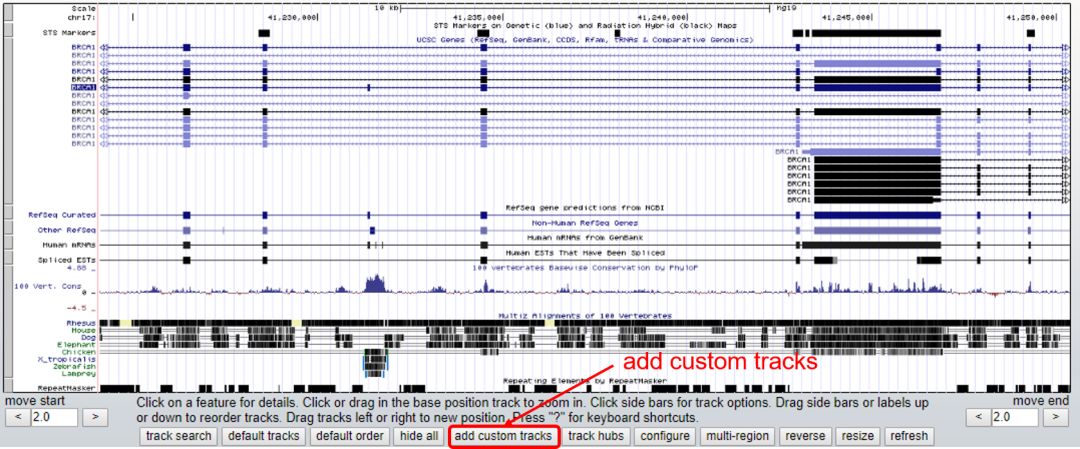
图 8. 利用 add custom tracks 对数据进行可视化
这个部分你点击它,上面有它支持的文件格式。如果你用常规的 RNA-seq 或其他 seq 的处理模式,都可以转换成这样格式的文件。但如果我关注的基因很少,没有向分析人员要到这种格式的文件,你也可以按照这类的格式的文件,自己做成这样的格式,也可以上传。
BLAT (BLAST-like alignment tool)
第二个板块,介绍一下 BLAT 的功能,它类似于 NCBI 里的 BLAST,但稍有不同。很多人都混淆着用,但可能会用错。对于 DNA 的序列,BLAT 至少要 40 个碱基(最新版的网页中显示,至少需要 25 个碱基)。对于蛋白序列,至少要 20 个氨基酸序列。低于这个数量,它的结果是不稳定的,或是你拿到了假的分析结果,有可能导致后面一连串的生物实验验证不出来。
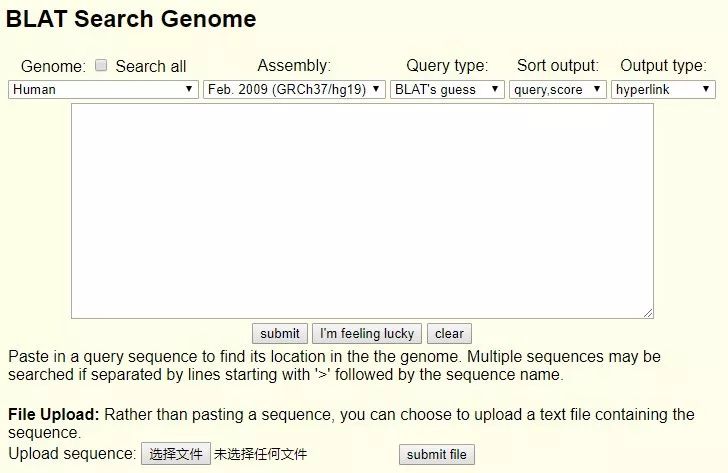
图 9. BLAT 的使用界面
对于一段给定的氨基酸序列,选好了版本和物种,如果你什么都不选,它有一个初始的阈值,你就 submit 一下。如果序列很多,你可以上传一个序列文件。其结果类似于 BLAST,显示哪些部分跟什么(序列)很像,每一个部分的打分也都会在下面有所显示;后面还有整个的匹配程度,显示哪些序列匹配哪些不匹配。
Table Browser
ENCODE 计划里面的数据我怎么下载呢?这个部分我们会经常用到 Table Browser。
Table Browser 简介:
1. 提供了访问数据库的便利入口;
2. 用文本形式来获取存储在 Genome Browser 数据库中的基因组汇编和注释数据;
3. 为整条染色体或一些特定的序列获取 DNA 序列信息或注释信息;
4. 用特定的条件对输出结果进行筛选;
5. 创建自己的路径,并且在 Genome Browser 中可视化地显示出来;
6. 整合多重查询,并为其产生相关的输出;
7. 为选定的数据集提供基本的统计结果;
8. 显示一个数据表格的详细情况,并且列出在数据库中与其相关的所有表格;
9. 根据其它应用程序和数据库的要求,对输出结果进行格式化。
总体而言,Table Browser 可以帮助大家进行数据下载的筛选;如果你不进行筛选,它有一个 download 的部分,download 的数据是不可筛选的。如果你是做特别的 de novo 的东西的时候,可能要下载全数据。但是如果你只关心 repeats,其他的区域我不想要,我就可以用这个功能去过滤。怎么过滤呢?
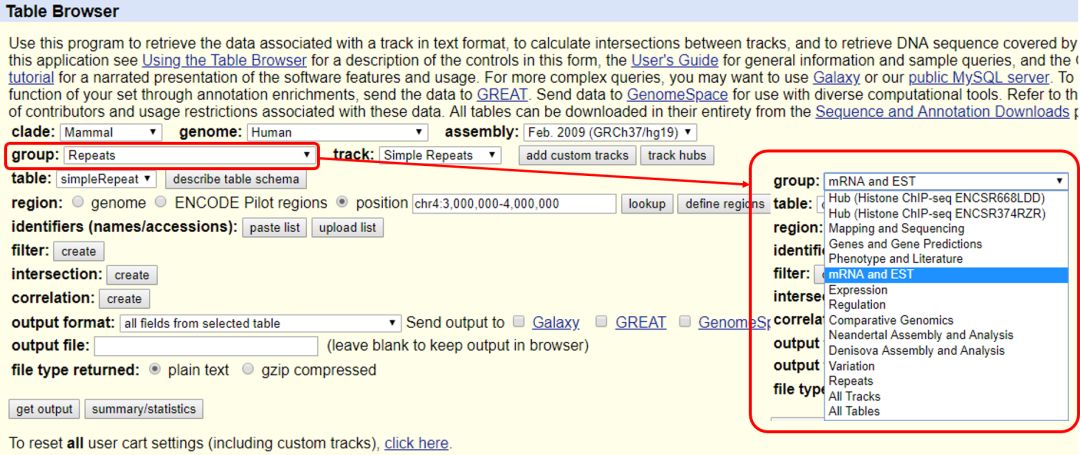
图 10. Table Browser 的页面
我们看 group 中有很多的条目,如果所有数据都要,你要把所有的 tracks,所有的 table 都给拿出来,但这个要很久。对于特定分析来说,你用其中的一部分数据就 OK 了。举个例子,下载从第四号染色体开始简单重复八次以上的人类序列。我们怎么从 Table Browser 里把这条信息或者这个 table 拽出来呢?Table Browser 点进去以后,在 group 选项中选择 Repeats,在 track 中选择 simpleRepeat,region 中的 position 修改为 chr4:3000000-4000000,在 filter 选项中点击 creat 按键,点一下就会出现很多更细节的选项。
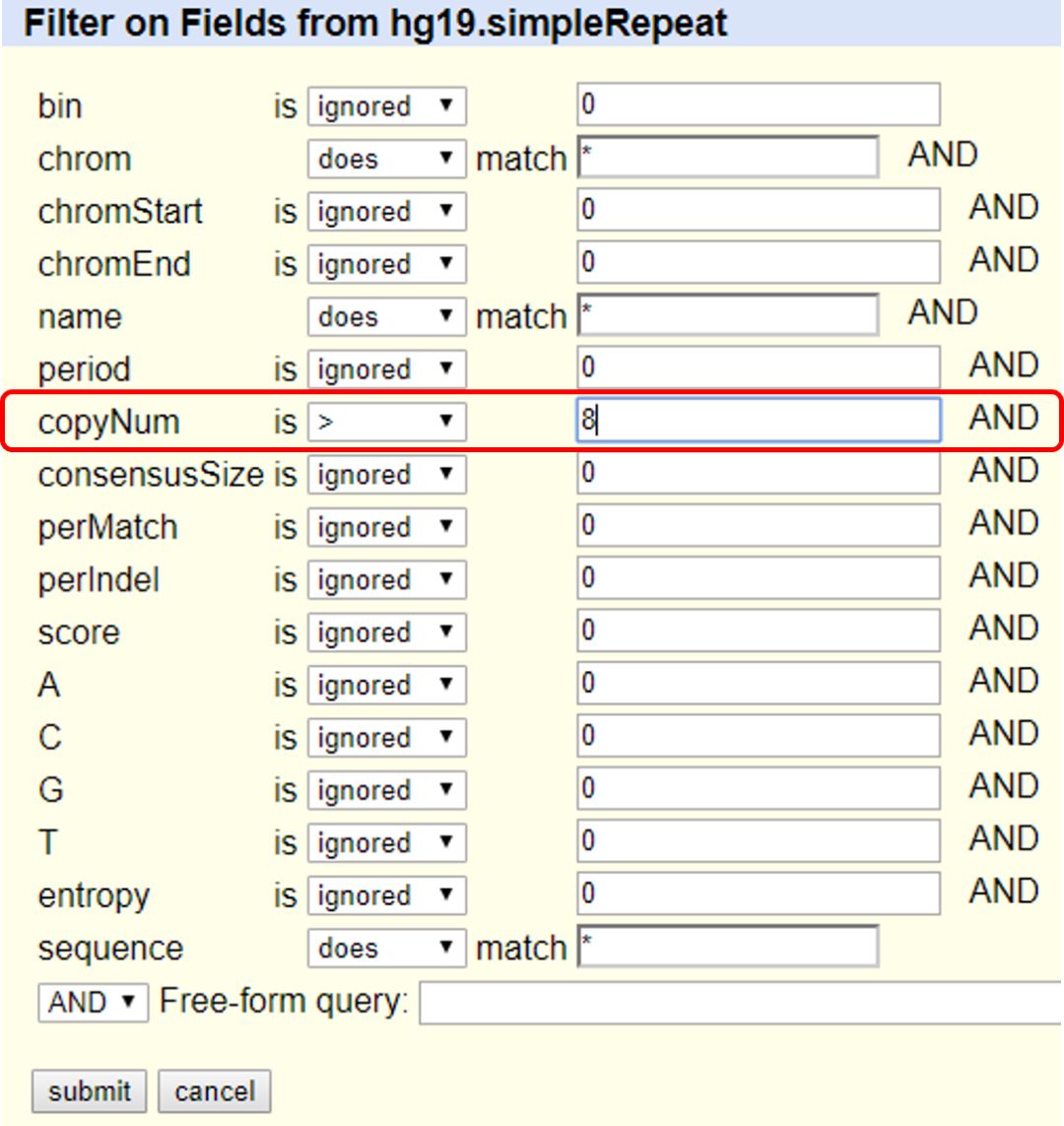
图 11. Table Browser 的 filter 选项中的详细参数
正如我们需求的,它的重复要在 8 次以上,copyNum 选项设置为大于 8 次,点击 submit,此时会返回上一步的界面,注意此时的 filter 选项已经变成了 edit 和 clear,edit 可对此前设置的参数进行修改,clear 则可清除此前设置的参数。在该页面继续点击下方的 get output,此时输入的结果就如下图。
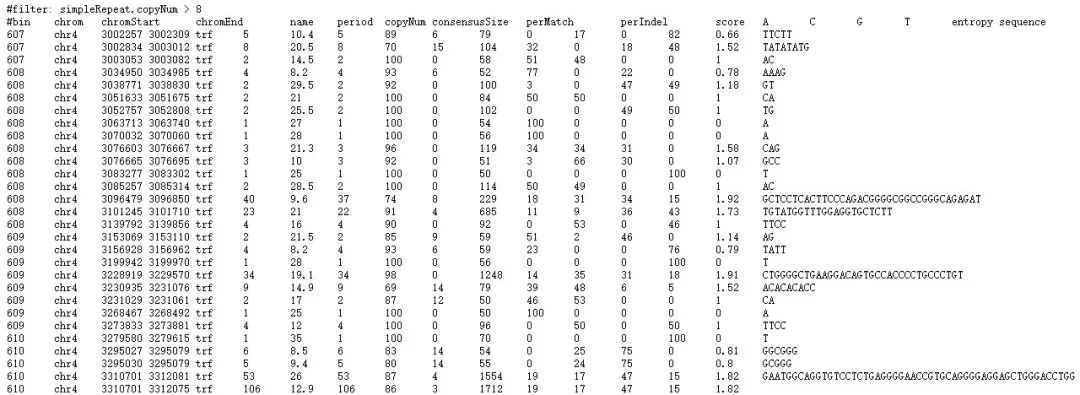
图 12. 第四号染色体重复八次以上的人类序列
图中包括它的位置、染色体的一些详细信息,后面它的一些序列信息,在这个表格里头都是有很详细的记载,你也可以直接把它下载或者复制出来都行。有些人可能不太喜欢用 download,如果数据少的话,你复制出来格式也不会变。
Gene Sorter
经常会有人有这样的问题,我们组里一直研究某个基因,研究到我这以后,我都没法做功能了,别人已经做的很清楚了,我该怎么办,后面我该怎么走?要不我做个基因互作?那我该怎么找和这个基因具有关联的基因呢?还有比如其他人研究的是人类的这个基因的功能,而我关注其他物种里的同源基因,这个时候我该怎么办?Gene Sorter 可以满足我们的这些需求。
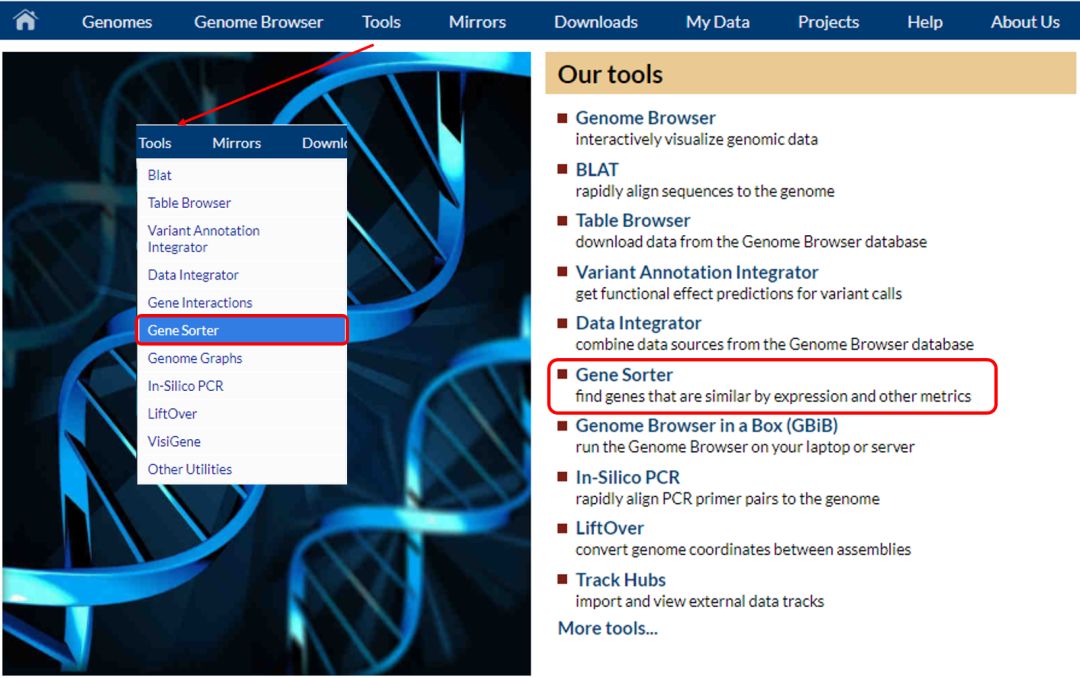
图 13. Gene Sorter 的两种进入方式
Gene Sorter 简介:
● UCSC Gene Sorter 是一个用来开发基因家族和基因间关系的一个非常优秀的资源。这个工具显示了与所选基因组相关的其他基因组列表。
● 通过这个工具可以找到蛋白水平的的相似性、基因表达谱的相似性或是基因组的相似性。
Gene Sorter 点开以后,可以看到有基因表达的信息(通过 Expression 进行 sort)、同源性的信息(通过 Protein Homology-BLASTP 进行 sort)、还有基因间的距离(通过 Gene Distance 进行 sort)。我们要搞清楚某个基因被注释到哪个功能里,为什么说它被注释到哪个功能里?因为基因 ontology 是一个像树根一样多层级结构,同一个基因有可能在基因 ontology 的注释里出现在多个功能上。它有点类似于我们人体的系统,几大系统下面有组织,下面分得更细,基因它有可能出现在多个功能节点上。这个功能节点越接近底层的部分,功能就越细致;越在上游的部分,功能就越含糊。比如说转录调控是一个功能,正向转录调控就是它下面层级的一个节点了。
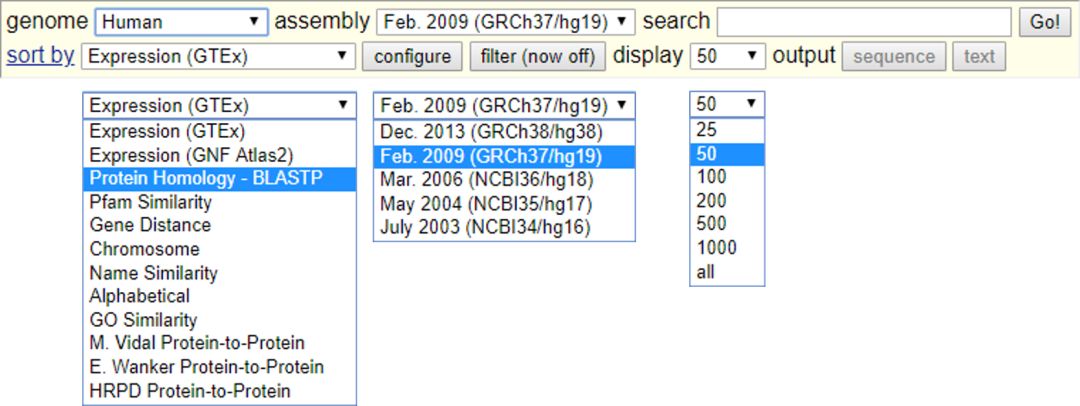
图 14. Gene Sorter 的具体参数
如果关注和某个基因功能相关的基因,可以选择 Go Similarity 的选项。如果关注蛋白质,有哪些基因和我这个基因相互作用,根据不同的算法背景,有这几种预测的一些方式。如果要做人类的蛋白,HRPD(Human Protein Reference Database)Protein-to-Protein 就可以了,其他两个数据库(M. Vidal Protein-to-Protein & E. Wanker Protein-to-Protein)还涉及到其他物种的。
如果选择 Protein Homology-BLASTP,就会输出跟这个基因同源的相关蛋白。configure 为结果显示的选项,决定了结果的呈现方式;如果输出的结果很多,你也可以用 filter(now off)选项过滤掉一部分。以 BRCA1 基因为例,输入 BRCA1 后,点击 go! 输出过滤后的结果,见下图 15。如果点进去其中任何一个基因,你会看到一张表,上面有一堆信息。应该先筛选有用的信息,再去仔细地看。

图 15. 与 BRCA1 同源的基因
In Silico PCR
最后一个板块,UCSC 也提供了一个 PCR 的引物搜索的工具——In Silico PCR,即电子 PCR 或模拟 PCR,可用一组序列作为 PCR 引物来搜索数据库,返回相关的序列。
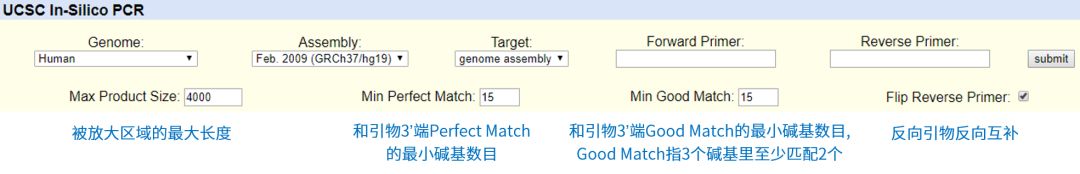
图 16. In-Silico PCR 的页面
以正向引物 GCCACAGTGCTCCGGA,反向引物 AAATGATCAGTAATCT 为例,输出结果如下图 17,下面还显示了引物的溶解温度。
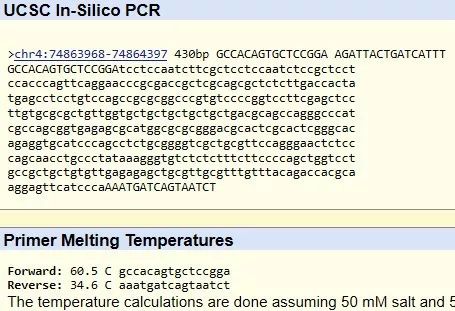
图 17. UCSC In-Silico PCR 的结果页面
Roadmap server
http://epigenomegateway.wustl.edu/browser/
上面我们讲到了 ENCODE 计划里数据的下载、可视化和筛选,在表观遗传里面还有另一大计划——Roadmap 计划,它倾向于检测人类、动植物正常的组织样本。如果你的研究需要用到 control 的话,这是一个很好的资源。这里面包括表达数据和各种类型的 ChIP-seq 的 data,包括甲基化的修饰,乙酰化的修饰,你都可以找到。特别是有些细胞系的,如果它测过了,你完全可以拿它的数据来用作为你的 control data。它的缺点也在于它只测了正常样本。
Roadmap server 是我们 2015 年 10 月王艇老师的实验室开发的,这个 Browser 的部分就是专门为我们那次会议开发的。(由于网页版本更新,新版本页面内容与旧版本有较大出入,不过具体细节功能几乎没有变化。只是进入操作上略有改动,可通过 The ‘old’ Browser 直接进入旧网页。)
图 18. WashU EpiGenome Browser 的页面
关于 WashU EpiGenome Browser 的使用教程见下面链接,以下不具体展开。
1. WashU EpiGenome Browser Tutorial
http://epigenomegateway.wustl.edu/support/2019CityOfHope/2019CityOfHope_Tutorial.pdf
2. Handout for WashU EpiGenome Browser Tutorial:
http://epigenomegateway.wustl.edu/support/HandoutforWashUEpigenomeBrowserdemo.pdf
3. Roadmap Epigenomics Workshop
https://www.genome.gov/Pages/Research/ENCODE/Tutorials/2016-03-17_SoT_WashU_Epigenome_Browser_Tutorial_Wang.pdf
4. WashU Epigenome Browser update 2019

图 19. WashU Epigenome Browser 的最新版本
PART 2: RNA-seq 数据的生物信息处理
我想分享给大家做 RNA-seq 分析的基本步骤、关键步骤、主流方法和较新的方法。我们实验室的科研模式是倾向于先有想法,然后通过已发表的数据去验证想法是否正确,验证预期结果是否有效,这时会涉及到如何使用数据库提供的或是别人已经发表的数据来验证自己的想法。
ENCODE 和 Roadmap 计划中的大部分数据,是可以在 NCBI 的 SRA 部分下载得到。在此我们举个例子,告诉大家如何在 NCBI 中下载得到一些公共数据?如何去找你关注的癌症或其他疾病的数据。NCBI 的主页大家肯定经常用,比如 PubMed。在此,我们不选择 PubMed,选择 SRA。你研究或关心什么疾病,就输入什么关键词,比如 lung cancer。
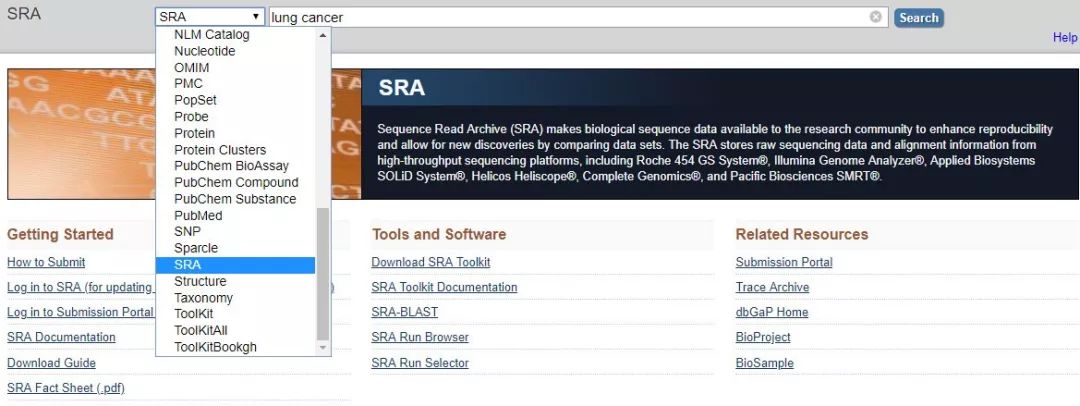
图 20. NCBI SRA 的使用页面
搜索之后,类似于 PubMed 检索,会出现很多的条目。下图 21 右边的框中显示了所有的数据,但并不是所有的数据都提供下载。对于 public 部分,原始数据和处理后的数据都可以下载。如果是 control 状态,有可能是文章已经 submit 了,杂志要求数据上传,reviewer 要看我的数据,作者在上传数据后会设置公开日期,比如我认为文章一年后会被发表,就把实验数据的 control 日期设在了一年之后。这样的数据只能看,无法下载,能下载的数据只是 public 部分。
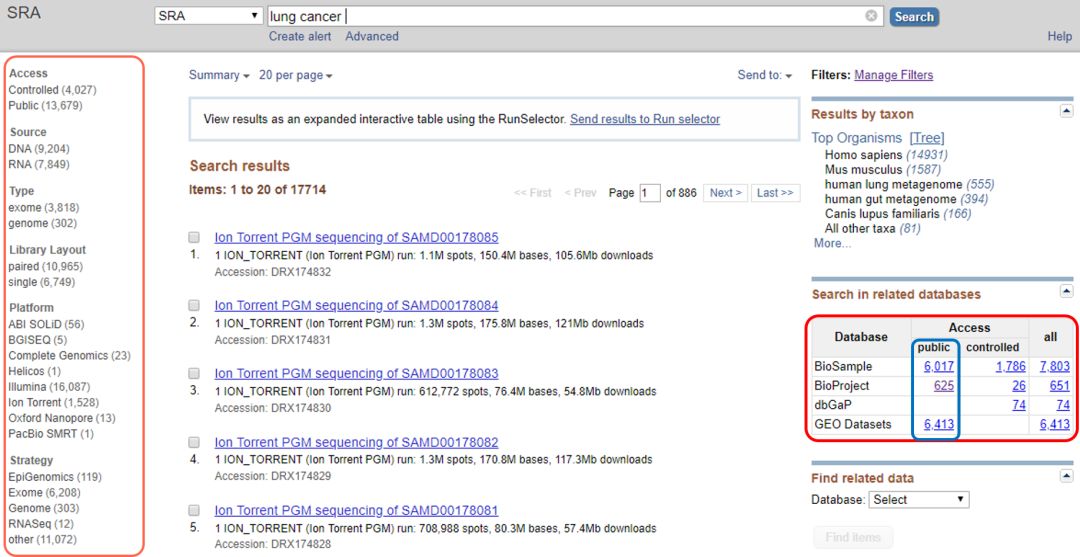
图 21. SRA 中 lung cancer 相关数据概览
也可在左侧对数据进行筛选,随后筛选物种 Homo sapiens,继续选择 GEO Datasets,结果仍然像 PubMed 一样,出现很多条目的信息。对于这个数据库来说,我们只能一个个地打开,然后快速扫一眼。如果你对数据很熟,通过对下方描述信息的简单阅读就可以很快过滤到所需信息,如果你不熟,你就都看一看。
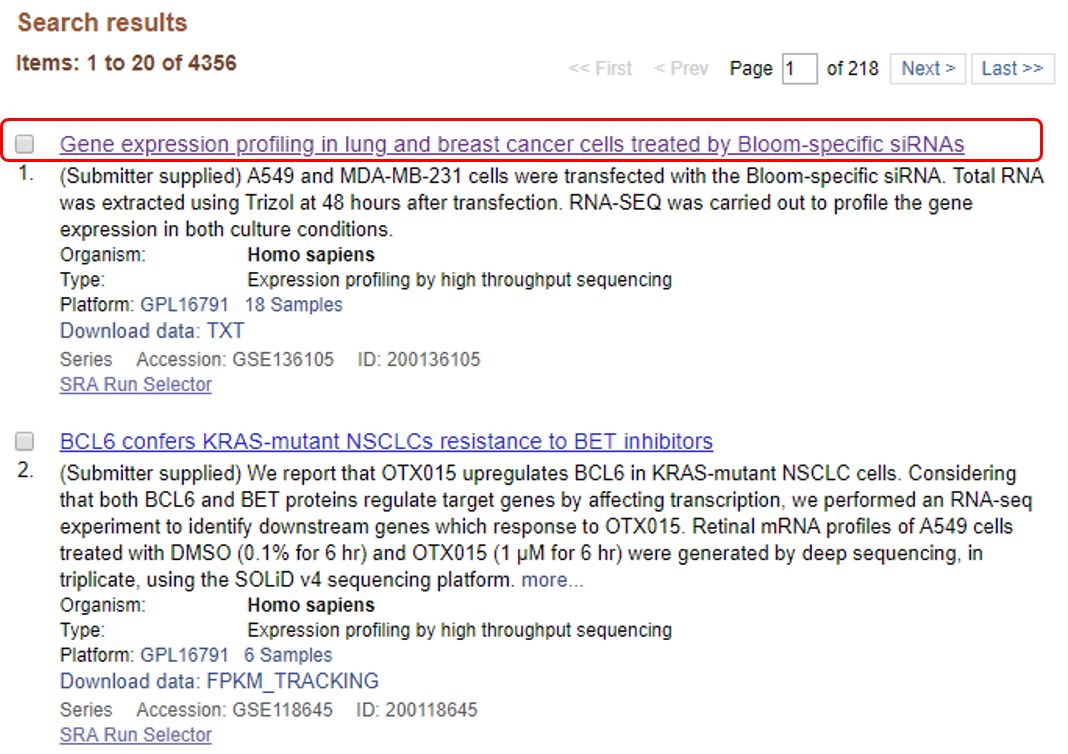
图 22. SRA 中 lung cancer 相关的 GEO Datasets 概览
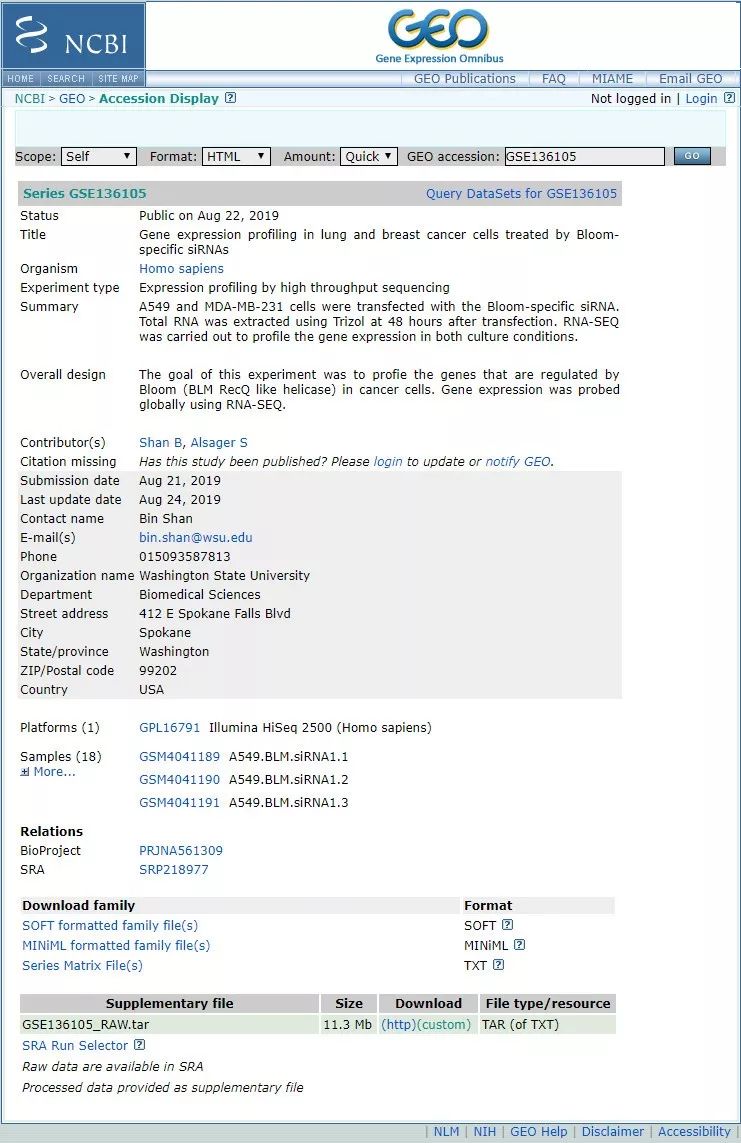
图 23. GEO 数据概览
进入界面之后,首先检查物种是否与我们关注的物种一致,然后快速浏览 Summary 和 Overall design 部分的内容,这个部分会说明他的实验是如何做的,样品是怎么处理的,看一下与你的预期是否一致。在 Platforms 部分会告知平台,现在大部分 Seq 数据都是 Illumina 平台,更早的还有像 Affymetrix、芯片等。Samples 显示样本信息,一般是简称,比如这里 control 有 6 个,case 有 12 个,一共 18 个样本,可下载相关描述文件对样本进行鉴别。下面的 SRA 部分,点击相应的序列号 SRP218977,可以看到 18 个样本的列表,点击第 1 个 GSM4041206: MB231.CTL.3; Homo sapiens; RNA-Seq 进入详细的页面。

图 24. GEO 数据中样本的详细信息界面
在最底部我们可以看到有一个 SRR 的编号,这个数据很大,有几个 G,通常我们不会在 PC 机上进行分析,而是在 Linux 平台上。点击 SRR 序列号,会有更详细的信息。
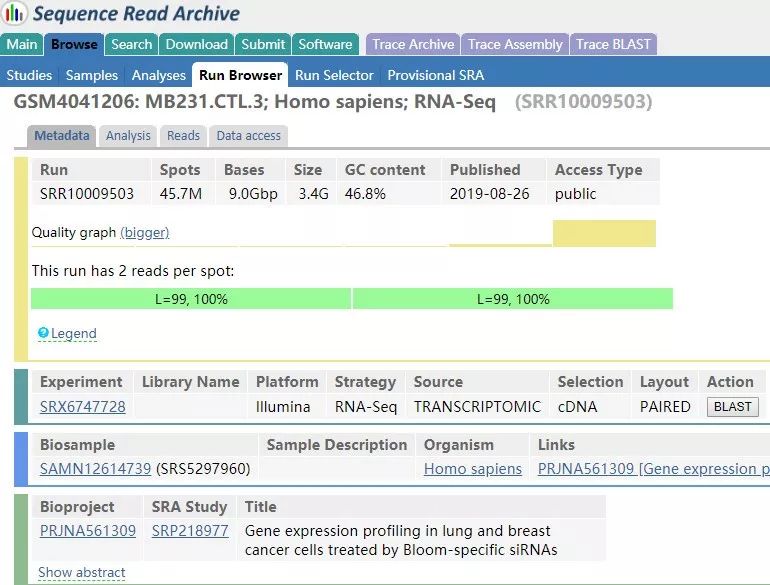
图 25. SRR10009503 数据的详细信息
SRA 的数据有自己下载工具。在 NCBI 的主页上有专门的下载工具 Aspera,对于它这种压缩格式的下载速度还是可以的。
图 26. NCBI 中的下载工具 Aspera
下载到本地之后,不能直接打开的,直接打开是乱码。因为它是一种压缩格式,而不是可视化格式。你要使用 fastq-dump 工具,把压缩格式转成 fastq 格式,这是我们从公司测出来数据的标准格式。以下以 SRR518622 为例进行演示。

图 27. 使用 fastq-dump 将压缩格式转化为 fastq 格式
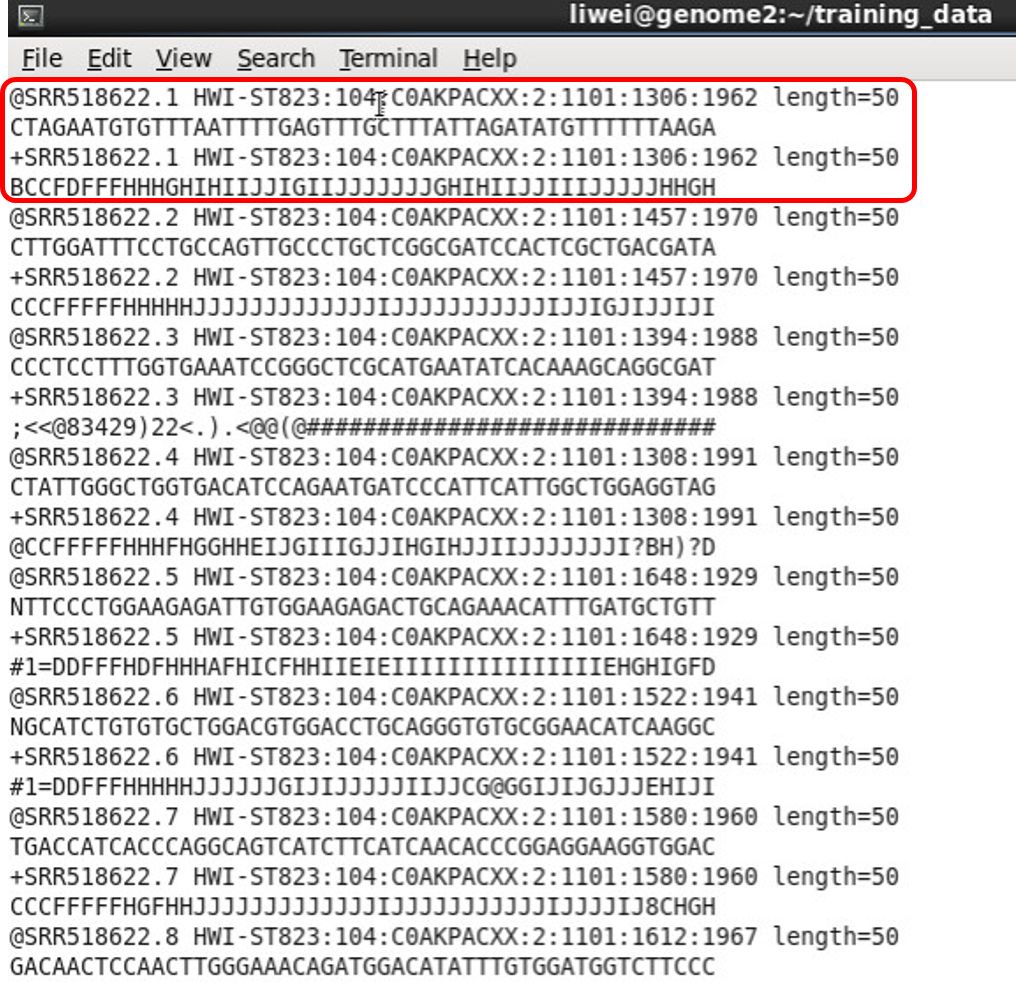
图 28. fastq 数据格式
fastq 格式是什么样子的呢?一个 fastq 文件有很多行,每 4 行代表一个 reads,就是你测到的一个 DNA 片段。第一行和第三行是描述,通常我们每套数据的第一行都是类似的,但是它们里面包含的具体信息不一样,有些包括整个长度、barcode 信息,每一个 read 最后都要有 barcode 才能分出来。第二行是具体的序列信息,最后一行是碱基的质量信息。这就是后续做 QC (Quality Control)的时候的评价,比如都是 100bp 的序列,我们怎么评估它测得好不好。
通常公司把数据给你,或者我们自己下载了数据之后,我们首先要评估一下 QC,或者说评价一下数据质量好不好,那么怎么评估数据质量好不好呢?这里有一个使用起来相对简单的工具——fastq QC,但它不是最好的工具,只是它的操作比较简单。如果说你有 Linux 系统的话,可以直接把你关注的数据选进去就行了。
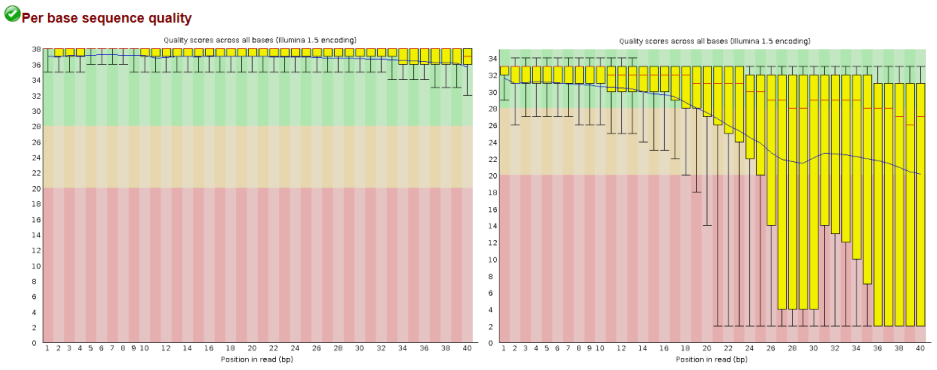
图 29. fast QC 产生的数据质量报告
这就是它的结果报告,主要包含三种颜色,绿色、黄色和红色。跟交通灯一样,绿色是 pass 状态,黄色是一个可忍受状态,红色说明这个质量非常差。比如我测序长度是 40bp,测了两个样本,然后公司给我结果,我就做了两张 QC 的图来评价这 40bp 里面每一个位置的测序质量怎么样,就这样看的话,左边的变异更小,所以更好。
如果你细心的话也会发现,对应的区域的颜色也可以说明,从下到上依次为红色、黄色和绿色,如果大部分数据都处于绿色,就说明质量还可以,拿到数据是可以付款的。但是如果说变异是右边这样的,绿色也有,黄色也有,红色也有,这个时候你要考虑公司是不是测错了,你要找他 argue,有可能是他测序过程中出现了什么问题,使得你的数据呈现这种状态。当你在维护自己的权益,让公司帮你重测的时候,你应该拿出一些证据出来。
我们拿到数据之后,就要做 map。现在相对早的方式,像 bowtie、BWA 可能大家都会用,这里我们以 bowtie 为例。Mapping 是指把测到的数据向参考基因组拼接的过程,结果报告会显示,你的 reads 有多少 map 上了,然后会生成一个相应的文件,每一行是什么都会有注释。
还有我们现在处理 RNA-seq 的比较经典的方法是 tophat、tophat2。tophat2 除了把你的数据 map 到基因组上,同时还会告诉你它是不是存在一些 junction 的位置,做 splicing 的时候,需要看 junction 的位置是不是有缺失,哪些位置发生了缺失和插入,都有专门的文件来帮你预测。
Bowtie 的结果输出的 SAM 文件,为什么要强调 SAM 格式呢?因为 SAM 格式信息很多,你不管后续做什么分析,大部分数据都可以从 SAM 格式中抽提。Tophat 运用的 map 过程是 bowtie 的,你把数据 map 完了之后,可以生成一个 SAM,BEDtools 可以将 SAM 转成可以上传到 UCSC 上看的文件,如果你有自己的数据,处理完了之后就可以上传到 UCSC 上看,比如我感兴趣的位置是不是堆积了更多的 reads。如果你没有你自己的镜像,你可以注册一个账号,把数据上传到 UCSC 中去看。
刚才我们提到经典的 RNA-seq 的 map,会采取是 tophat2,这是目前用的比较多的。这篇文章 2016 年 5 月发表在 Nature Biotechnology 上,它开发了一种新的 RNA-seq 分析的算法。我们做 tophat 时会选取一个最小的长度,其中输入的参数有最小长度限制。比如说你有一个长度为 10 的 reads,它会以每三个为一段的规律,把 reads 断成几份,比如说 123、234、345,如果它的碱基长度是 10,就可以拿到 8 个 seed。每个 seed 就是你 map 之前的 anchor,先用这三个锚定到基因组上,然后再往后延伸。它是三个三个延伸,比原来 anchor 二十几个 bp 的精准度相对更高。大家感兴趣的话可以看一下这个 paper。

图 30. RNA-seq 分析新策略
文章做了二十多个测试数据,来评价数据准确率平均值的波动情况,对于 Kallisto 而言,准确度波动不是很大,相对稳定。在计算的时候,是基于哈希表的原理,把一个个种子先存在哈希表里,然后在哈希表里找。而且 Kallisto 的运行时间较快,如果大家用了 tophat2 发现整体 map 结果不太理想,可以试一下。
提取表达谱,我们用的比较多的是 cufflinks。用 cufflinks 提取时,每个基因是有指定的表达值的。有的数据显示的是 RPKM,有的是 FPKM,它们有什么差别呢?其实对于单端测序的数据来说,它们是一样的,如果你测的是 paired-end 的数据,也就是有可能一段区域里面有 paired-end 的两个位置的时候,RPKM 就相对不准了,R1 算一次,R2 又算一次。我们现在用的更多的是 FPKM。
RPKM 如何计算的呢?RPKM 是指 Reads Per Kilobase per Million mapped reads,即每百万 reads 中来自于某基因每千碱基长度的 reads 数。比如某个基因上有 1000 个 reads,总的 reads 有 100 万,外显子总长是 5kb,那么用公式算下来,RPKM = 1000 / (1 * 5) = 200,它的 RPKM 就是 200。如果你做的是双端测序,并且双端很好地 map 上了以后,它的数据就可能为原来的一半,但是这并不常见。
Part 3 : 表观遗传学常用软件及网站的介绍
接下来为大家分享一些表观遗传分析过程中常用的数据库,希望大家通过这个部分了解自己研究的领域有哪些资源可以用,以下数据库可能不是最好的,但都是比较常用的。
DNA 甲基化数据库
1. MethDB: http://www.methdb.de
2. PubMeth http://www.pubmeth.org
3. DaseMeth http://bioinfo.hrbmu.edu.cn/diseasemeth
组蛋白修饰数据库
1. HIstome: The Histone Infobase
http://www.actrec.gov.in/histome/
2. Human Histone Modification Database
http://bioinfo.hrbmu.edu.cn/hhmd
miRNA 相关数据库
1. miRBase http://www.mirbase.org/
2. miRWalk2 http://zmf.umm.uni-heidelberg.de/apps/zmf/mirwalk2/
3. miR2Disease http://www.mir2disease.org/
4. The human microRNA disease database
http://www.cuilab.cn/hmdd
5. Mir2subpathway
http://210.46.80.7:8080/miR2Subpath/
6. Transcriptional factor and miRNA regulatory cascade
http://210.46.85.180:8080/TMREC/
LncRNA 相关数据库
1. LNCipedia database https://lncipedia.org/
2. TheLncRNA and Disease Database
http://www.cuilab.cn/lncrnadisease
3. NONCODE http://www.noncode.org/
本文在有限的篇幅里对 UCSC Genome Browser 的使用、Roadmap 数据库、RNA-seq 数据的处理流程以及表观遗传领域常用的数据库进行了简要介绍。但实际上,任何一个数据库都可以单独再写一篇教程进行详细解读,工具的使用和熟悉还需要大家结合自己的实际研究进行深入探索!
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言












#遗传学#
41
对我来说很好哇
100
正在需求中,,
115