脓毒症亚型:进一步认识理解脓毒症的一种框架
2023-02-09 MedSci原创 MedSci原创 发表于上海
脓毒症被定义为感染引起失调的宿主反应所导致的致命性器官功能不全。它每年影响全世界大约5000万人,并且通常是致命的,即使及时应用循证指南也是如此。


中南大学湘雅医院 刘薇/艾宇航 译
中山大学附属第一医院 吴健锋 审
北京大学人民医院 赵慧颖 / 贵黔国际总医院 马朋林 述评
中南大学湘雅医院 艾宇航 述评
摘要:脓毒症被定义为感染引起失调的宿主反应所导致的致命性器官功能不全。它每年影响全世界大约5000万人,并且通常是致命的,即使及时应用循证指南也是如此。在过去的20年里,许多随机试验测试了脓毒症的治疗方法,但大多数无益。这可能因为脓毒症是一种异质性综合征,其特点是具有多种临床和生物学特征。然而,这些特征的组合可能会导致出现先前未被识别的或具有不同预后风险和对特定治疗反应的“亚型”。随着识别脓毒症亚型的努力变得越来越普遍,出现了许多悬而未决的问题和挑战。
包括:1)脓毒症亚型的语义基础,2)亚型的概念性目标,3)关注研究设计、数据源和统计方法,4)新兴数据类型的作用,以及5)如何确定亚型是否代表其“真实”性。我们探讨这些问题,并为更广泛的脓毒症亚型研究提出一个框架。该框架旨在帮助理解和解释脓毒症亚型、提供解释不同方法学产生的亚型的机制,并指导临床医生如何在床旁监测中识别脓毒症亚型。
关键词:异质性;表型;脓毒症;亚型
脓毒症是一种广泛异质性综合征,最近被定义为感染引起失调的宿主反应所导致的
致命性器官功能不全[1]。每年,全世界有近5000万脓毒症患者,病死率从5%~40%不等[2,3]。减少脓毒症患者死亡和不良结局的压力越来越大。然而,支持性治疗和新治疗措施的试验一直令人失望,这表明临床脓毒症的内在复杂性需要更复杂的方法对患者进行分层[4]。最近在更大范围的脓毒症中确定组群的努力带来希望,亚型或具有共同特征的患者采取针对性治疗将产生积极的疗效。这些治疗与病理生理改变一致,因此更具个体化。
一提到治疗方法明确的脓毒症患者,临床医生脑海中浮现一些原型,比如确诊为肺炎球菌肺炎的多病老年患者,然而,这只是众多脓毒症患者中的少数[5,6]。因此,人们基于不同的目标提出了各种方法将脓毒症患者分类(表1)。许多区分亚型的方法并非绝对,亦无对错,因而,我们建议在不同目的的研究设计中衍生的脓毒症亚型可能对患者监护起到补充作用,只要它们之间的相关性能得到充分理解。在这篇文章中,我们讨论了临床医生、研究人员、试验者和卫生政策制定者面临与成人和小儿脓毒症亚型相关的挑战。
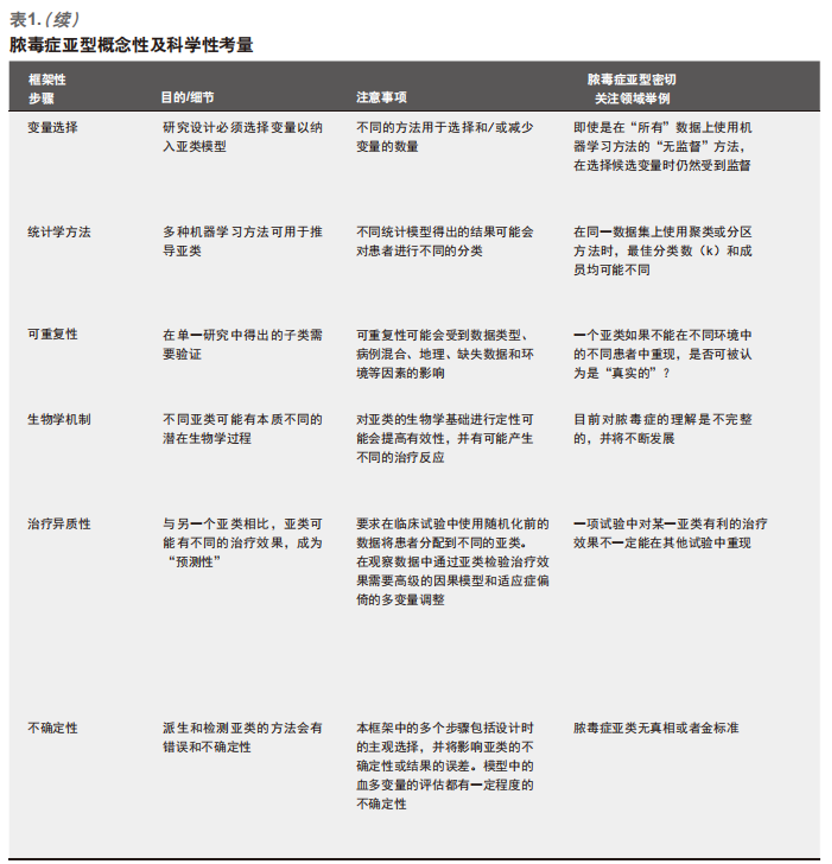
为何难以建立脓毒症亚型?
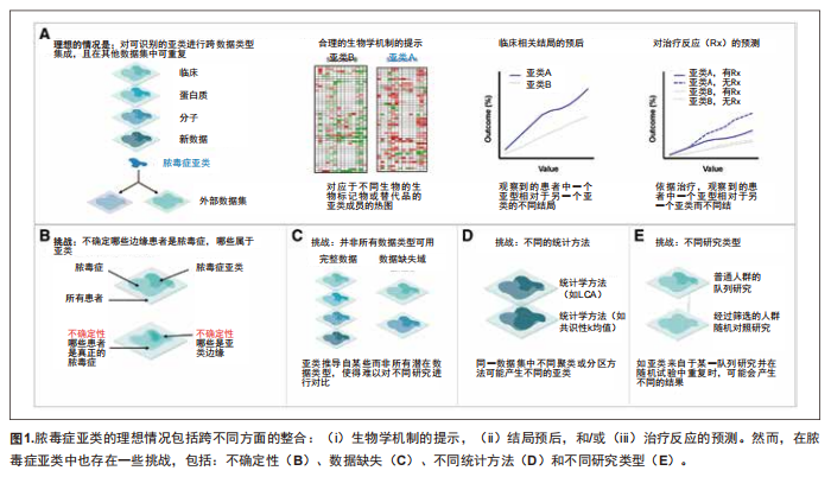
疾病分类是日常临床实践中的一个固有步骤,尤其是在重症状况下[7]。我们经常会考虑一种疾病的存在或不存在,倾向于做出二元决策来支持治疗(例如,开始即使用抗生素治疗感染)。对于大多数情况,这种分离的、明确的决定是无法实现的。尽管分类背后的“真实”可能很复杂,但它仍然是理解生物机制和治疗反应的动力,也是沟通和决策制定的关键。当主要疾病脓毒症诊断本身发生变化和不确定时,脓毒症亚型的推出就会面临挑战(图1)[1]。
以往有研究表明临床医生不认为脓毒症的诊断是客观的[8],修订后的脓毒症标准所识别的患者与先前的标准中标记为脓毒症的患者不同(例如,“Sepsis-2”)[9-11]。
当对术语或语义缺乏一致意见时,问题就会更复杂。例如,传统的脓毒症分类方法通常按疾病严重程度分层,而没有按病理生理学进行区分。未来的亚型可能会考虑这些因素的组合。当脓毒症、感染和急性器官功能障碍的组成部分本身具有不明确的“边界”时,就会出现更多的不确定性[12]。
慢性器官功能障碍(如肾脏疾病)也是临床医生和研究人员面临的挑战,他们需要区分新的变化是感染,还是慢性疾病的发展,亦或与两者的组合有关[13]。这些决策的边界模糊会影响亚型的可信度和可重复性。
悬而未决的问题如下:
1)在定义亚型时,我们如何考虑不确定性和临界患者?
2)亚型是没有关联的吗?
将脓毒症分为亚型时的目标是什么?
脓毒症的预后仍然令人沮丧[14]。 脓毒症的新治疗措施应减少可避免的死亡并改善脓毒症幸存者的生活。 然而,哪些患者是可避免死亡的?哪些幸存者的生活能够得到改善?且为什么能够得到改善? 一种方法是根据预后确定分类,这种方法根据患者因脓毒症死亡或其他靶向不良预后的可能性对患者的风险进行分层[15]。例如,根据先前存在的合并症或多器官功能障碍来区分患者,可以将一组具有高死亡风险的患者从所研究的疾病人群中区分出来[13]。 此外,由于导致脓毒症死亡的共同因素的复杂相互作用,使用预后富集的临床试验尚未确定哪些治疗是有效的[15,16]。
另一种方法是根据预测来确定组,根据患者对基于潜在生物学机制的特定治疗做出反应的可能性进行分组,而不管预后如何。预测富集不应与预后富集混为一谈,尽管存在重叠的情况[17]。比如,急性呼吸窘迫综合征(acute respiratory distresssyndrome,ARDS)中的严重低氧血症,严重低氧血症既可以预测又可预后,因为它可能与更高的死亡率及更多的患者从俯卧位中获益有关[17]。
尽管有望基于生物学上可信的途径来指导治疗,但迄今为止,这些尝试还没有在脓毒症中产生有效的治疗方法,也没有在随机对照试验(randomized controlled trial,RCT) 中得到充分验证[18]。展望未来,我们预测新的计算方法将使用综合临床、分子和组学数据的数据库为亚型提供信息。随机试验中的前瞻性验证,用于预后和/或预测性富集[19,20],将是确保亚型分类方法实用、揭示独特的病理学并在不同场景实现治疗目标的关键。
悬而未决的问题如下:
1)同时期所有随机试验都应该包括预后性或预测性富集吗?

我们将使用哪些词汇?
文字很重要。选择正确的语言对脓毒症进行分类,使临床医生能够对患者(高危患者)进行分层、告知(潜在原因是什么)并及时采取(治疗措施)。脓毒症的认识论是令人困惑的,这是因为不同的模式识别技术用于衍生脓毒症亚类。研究人员采用的技术从经验方法到机器学习,新的术语不可避免地被引入,这些术语很少与以前的工作协调一致(图2)。
在过去的20年里,诸如“亚组”之类的术语已被用于根据感染源(例如,社区获得性肺炎与呼吸机相关性肺炎)、器官/组织(脑膜炎与尿路感染)或生物学特征(高炎症性与低炎症性脓毒症)来调整患者分类。
现在,数据驱动法以不同的方式对脓毒症进行分类[例如,“脓毒症的分子诊断和风险分层(molecular diagnosis and risk stratification of sepsis,MARS2)”或“δ临床表型”][6,19,21]并将这些组称为亚型、内型、临床表型或亚表型。 术语可互换使用且不一致,研究人员缺乏共识。已经有文献讨论和总结了关于术语的争论[17]。在这篇文章中,我们使用“亚型”来表示一组患者,而不考虑潜在的分组方法。
悬而未决的问题如下:
1)除了“亚型”之外,是否有统一的术语来描述脓毒症亚组?
2)哪些步骤将减少临床医生和研究人员之间因语义不同所导致的误解?
我们应使用什么数据源?
为了确定脓毒症亚型,研究人员可以使用许多数据源(图2)。用于衍生、复制或探索新的脓毒症亚型的数据库的选择可 能会影响研究结果。例如,多中心随机对照试验数据可能来自不同地区登记的多个中心,具有较高的内部有效性,并且可以通过利用随机化建立因果关系。在RCT的病例报告表中,缺失数据不太常见,因此RCT在治疗分配和依从性方面有很高的自信度[16,22]。然而,脓毒症中的大多数RCT都排除了可能会限制亚型的外部有效性的因素,例如共病、预期寿命或疾病严重程度。
观察性队列研究可以探索脓毒症亚型。使用电子健康记录(electronic health record,EHR)队列数据可以灵活适应不同的脓毒症标准、发病时间、多种病原体,并且通常每天包含多个数据点[6]。观察性队列研究也可以大规模扩展到低中收入国家[3]。样本量可能比RCT高很多倍,从而为统计算法提供了更多的权重。尽管观察性队列研究通常可在患者、医院或系统层面进行推广,但它们也可能包含缺失数据、异常值、临床实践随时间的变化,以及被未知变量混淆[5]。更严格的前瞻性队列研究可以克服其中的一些问题,但历来难以获得资金,并且缺乏评估因果效应的内部有效性。
最后,结合队列研究和RCT的数据可以收集到更多的信息。 例如,急诊监护中的脓毒症内表型(the Sepsis ENdotyping in Emergency Care,SENECA)研究报告了来自多个EHR队列的脓毒症表型,这些队列在5年内累积了来自12个中心的患者[19]。然后,在具有不同脓毒症类型的历史队列中复制表型并应用3项RCT研究[16,22,23]。从概念上讲,这种方法“开始广泛”将脓毒症的异质性最大化,再进行分类,然后试图在更高策划的数据中“验证”或重现这些发现。最后,如果提出脓毒症亚型的方法能够反映亚型之间生物学的真正差异,那么研究结果将适用于不同的数据源、脓毒症的定义、治疗和地域。
悬而未决的问题如下:
1)相较于单一数据,联合观察数据和试验数据是否可以更好地模拟亚型治疗效果?
2)如果仅根据一项试验的分析报告,亚型治疗效果是否与临床医生相关?
基于临床或转录组学数据定义脓毒症亚型
在探索脓毒症亚型时,有许多数据类型需要研究(图2)。其中最常用的是临床、蛋白质和/或转录组学数据。临床脓毒症亚型具有不同的实验室异常模式,有器官功能障碍,并且可以预测临床结局(在线表1,http://links.lww.com/CCM/G108)[19]。
然而,单独的临床数据提供的生物学机制信息有限,但测量宿主的生物标志物可以提示导致临床表型的潜在 生物学机制[19]。EHR中临床数据可能相对稀少,以自由文本形 式存在,难以分析,并且仅限于现成的测量数据,例如生命体征、药物和实验室检查[6]。然而,汇总数百万患者的数据有助于发现具有说服力的脓毒症亚型[19]。
另一种方法是使用代谢组学、蛋白质组学和最常见的转录组学来识别脓毒症亚型(在线表1http://links.lww.com/CCM/G108)。这种方法的关键是技术进步使人们对全血基因组表达和白细胞转录组有了详细的了解[24,25]。例如,脓毒症基因组学发展联盟和 MARS联盟分别使用全血基因表达来鉴定社区获得性肺炎、粪性腹膜炎及其导致的脓毒症亚型[21,26,27]。
尽管这些亚型的确切数量和组成各不相同,但共同因素仍然存在,例如与疾病严重程度和28天病死率的关联。这些特征是脓毒症特有的还是与脓毒症之外的疾病相关尚不清楚。然而,最近的证据表明,对不同类型的应激可能确实存在基本相似的反应。
在Xiao等人的一项研究中[28],不同的损伤机制、外伤、烧伤和内毒素血症在基因表达模式上具有相似性。此外,急性胰腺炎的亚型可能与ARDS的亚型相似[29]。因此,在脓毒症中,至少 有一些分子亚型可能会反映宿主对严重全身性损伤反应的一般特征。
为了在临床环境中实现分子亚型的应用前景,有些困难必须提前考虑。检测技术必须近乎实时地执行,成本合理,复杂度低,并且可重复。幸运的是,实时RNA、蛋白质或代谢物定量检测现在更加便宜易得了。
悬而未决的问题如下:
1)如何将分子亚型从研究扩展到临床实践?
2)哪些计算技术能够从综合数据(包括临床和组学来源)中获得亚型?
用于识别脓毒症亚型的新兴数据类型
现代分子技术已迅速扩展。在单个患者标本、单个时间点可以产生数十万的检测结果,为我们提供有关宿主基因组学、基因表达、蛋白质合成、代谢活动、微生物特征和功能以及治疗干预效果的信息[17]。 因此,当在多个时间点和潜在的多个位置进行检测时,评估宿主对感染和治疗的反应的信息会大量扩增。这些丰富的数据为我们提供了推进脓毒症亚型的机会,但也带来了未解决的方法学和分析方面的挑战。
从根本上说,新技术提供了对患者的额外量化检测。这些检测在概念上与其他检测(如WBC计数或肌酐)没有区别,每个检测结果都是一个生物学过程的标志,该过程可能具有或可能不具有临床重要性。
举例来说,微生物组学是脓毒症亚型的一个新兴数据:研究发现患者体内存有1.5 kg细菌、真菌、古生菌和病毒。微生 物组在脓毒症的病理生理学中具有多种潜在作用[30]:1)潜在的病原体储存库,2)代谢/内分泌器官(将肠道内容物转化为全身活性小分子),3)局部和全身炎症介质,或4)影响细菌易位和细菌产物的来源[31,32]。与健康受试者相比,因脓毒症接受治疗的患者肠道和呼吸道微生物群都发生了明显的改变[31,33,34]。在危重患者中,肺微生物群的变异与全身和肺泡炎症的变异相关[31,35],而肠道细菌多样性的降低已证明可以预测 有脓毒症风险的患者的菌血症和死亡率[36–38]。
悬而未决的问题如下:
1)微生物组等新兴数据类型将如何识别由不同病原体引起的脓毒症亚型?
2)新出现的数据类型(如危重疾病的生态失调)是否在脓毒症生物学中起因果作用,还是仅仅反映了一种附带现象
儿童亚型情况是怎样的?
在儿科患者中的研究也提出了脓毒症亚型。这些研究使用各种概念性的方法,或者是有监督的、基于知识的亚型分类,或是基于基因表达的分类。例如,Carcillo等人[39]发现来自风湿病的特征确定儿童亚型,这些亚型对白细胞介素受体拮抗剂治疗(如巨噬细胞激活综合征)能够预测疗效并与预后相关[40]。另一种方法是非监督性的、不了解现有知识和确定数据而派生的亚型。在 PICU 患者中,Wong 等人使用基因表达谱来发现转录不同的亚型,这些亚型可以解释小儿脓毒症的异质性[41]。
不同的基因表达转化为具有临床意义的蛋白质生物标志物,两者可用于预测儿科患者的高死亡率[40]。儿童脓毒症亚型是存在的,并与成年患者的基因表达有一些相似性[42],并且可能与休克的类固醇治疗是否有效也相关[43]。
年龄是识别和理解儿科脓毒症亚型又一独特的挑战。儿童的年龄跨度大,会导致很多的可变因素:免疫反应、疫苗暴露、不同的病原体和生命体征,这些因素在一个年龄段可能正常,但在另一个年龄段可能异常。这些数据为亚型的临床解释带来了挑战性,因为亚型的生物学判定是否与年龄相关或独立于年龄并不清楚。
悬而未决的问题如下:
1)儿童脓毒症亚型对治疗有反应吗?
2)将青少年与年幼儿童进行比较时,儿科脓毒症亚型是否相似?
时间的重要性
关于时间与脓毒症亚型的关系存在争议。由于患者的潜在基因型(如21三体),一些人认为疾病亚型可能是终生的。另一些人认为亚型受特定的疾病阶段、监护时间或单一时刻的限制。例如,成人脓毒症的SENECA研究源于在急诊科6小时内分析的脓毒症亚型[19]。这些数据包括可能影响治疗和随机试验资格的临床时间点。
然而,狭隘的“时间”观存在许多缺陷。首先,它仅限于在指定时间窗内可观察到的数据,可能会错过其他时刻的信息数据。其次,使用单个时刻派生的亚型所识别的患者可能会在病程中发生变化[27,44]。尽管事实如此,但患者从一个亚型演变为另一个亚型可能会使临床医生感到困惑,并降低对此类亚型的生物学基础的信心,甚至使靶向治疗难以实现。
另一方面,随着时间的推移,亚型的变化也可能反映疗效、损伤甚至是结局[44]。 早期时间点的评估并不反映后续治疗的效果,而在后期时间点的评估反映了原始损伤和宿主反应影响的净结果,所有这些都可以通过临床医生的行为所改变。在确定亚型时使用重复数据或将时间视为连续变量可以帮助解决其中的一些问题。
悬而未决的问题如下:
1)脓毒症亚型发生变化的频率如何?
2)不断变化的亚型是否反映了预后、治疗反应、生物学 或所有三种情况?
获得脓毒症亚型的统计方法
亚型的三个主要统计目标(图2)是对患者进行分组:1)具有相似变量,2)预后的差异或相似性,或 3)不同的治疗反应。我们针对这三个目标中的每一个都给出了不同建模策略的建议(表2)。目标1通常使用无监督学习方法,因为主要目标不涉及对特定结果的预测[6]。相比之下,当目标2或3是重点时,可以使用监督模型更直接地导出亚型。监督结果模型被给予一组患者特征作为输入,并以合适的最能准确预测测量结果的方式进行。这些模型可应用于预后富集的可用协变量中开发基于风险的分组。监督方法也可用于通过协变量相互作用寻找特定治疗可能有益或可能有害的亚型。
为了避免过度拟合并确保预测准确性,使用测试或验证方法至关重要。在这些统计方法和许多其他方法中,策略的选择应主要由研究目标、既定目标和可用资源决定,最终,模型应具有可解释性和合理性。

探索亚型的意义
当提出一个脓毒症亚型来,我们如何知道它是否有意义? 有效性的测量方法是主要的问题。亚型之间的区别和成员之间的相似可能不足以让怀疑论者相信一组脓毒症患者属于一个整体。
新的亚型可以通过以下步骤进行评估:1)生物学合理性,2)预测治疗反应的能力,以及3)在其他数据集中的一致性和可重复性。还可以根据在临床监护治疗和/或临床试验中实施的可行性来评估一个亚型,然而,重要的是要注意这不会影响亚型的合理性、可预测性、一致性或可重复性。
也有可能提出一个亚型的数据库处于关闭状态,由于亚型模型中使用的试验或生物样本的性质,无法在后续数据中进行测试。否定被提议的亚型没有明确的标准。人们可能会发现用于获得亚型的设计或统计方法中的致命缺陷、对所研究的较大人群的错误分类问题、基础数据集的问题,甚至不可重复性。
识别同义组可能更容易,关键是包含定义潜在重叠类别所需的临床、生物或分子数据的数据集,如基因表达亚型所证明的那样,例如从其它疾病中识别SRS和MARS [21,26,45]。
亚型的临床应用?
将脓毒症亚型转化为临床诊断和/或治疗指导工具是实现个体化或“精准”医学目标的关键步骤。这一转变不可或缺的是将亚型嵌入到常规临床监护中——使用EHR。这样的过程可能需要以下信息学解决方案:
1)设定一组用于亚型分配的临床变量,
2)促使临床医生进行实验室检测或留取生物学样本(如果尚未获得)。
单个临床特征、基因或蛋白质生物标志物不太可能定义一个亚型,而EHR可以帮助管理整合多种数据[46]。除了亚 型识别和预警之外,治疗反应亚型还可以通过有针对性的临床 决策支持影响临床监护治疗。有研究使用强化学习算法[47],可以做到在患者进入某一亚型或疾病状态时提示治疗措施。最后,在一种“边做边学”的方法[48]中,亚型可以在指导脓毒症治疗改进的同时进行自我细化。
发展方向
在临床广泛使用脓毒症亚型之前,考虑与目的、统计方法、数据来源、时间和真实性评估相关的许多问题是有帮助的。为了预后或精确治疗,在单个数据识别迄今为止未被识别的一组疾病患者群是否足够?还是目标是真正个体化的策略?而不是“亚型”本身,也许我们应该寻找“临床相关的、非同义的、生物学上合理的、治疗敏感的和可重复的”亚型。一旦在有相应治疗的随机试验中进行测试,这些亚型不仅有可能为脓毒症的病理生理学提供信息,而且有可能在未来改善患者预后。
1 Department of Critical Care Medicine,The Clinical Research, Investigation, and Systems Modeling of Acute illness(CRISMA)Center, University of Pittsburgh School of Medicine,Pittsburgh,PA.
2 Anaesthesia, Critical Care, and Pain Medicine, Royal Infirmary of Edinburgh, Edinburgh, United Kingdom.
3 Division of Pulmonary and Critical Care Medicine, University of California San Francisco, San Francisco, CA.
4 Division of Pediatric Critical Care Medicine, Department of Critical Care Medicine, Children's Hospital of Pittsburgh, Pittsburgh,PA.
5 Department of Medicine, University of Pittsburgh, Pittsburgh,PA.
6 Department of Biostatistics, Graduate School of Public Health, University of Pittsburgh, Pittsburgh, PA.
7 Division of Pulmonary and Critical Care Medicine, Department of Medicine, University of Michigan, Ann Arbor, Ml.
8 Division of Anaesthetics, Pain Medicine, and Intensive Care,Department of Surgery and Cancer, Faculty of Medicine, Imperial College London, London, United Kingdom.
9 Wellcome Centre for Human Genetics, University of Oxford,Oxford, United Kingdom.
10 Department of Biostatistics, Vanderbilt University Medical Center,Nashville,TN.
11 Kaiser Permanente Division of Research, Oakland, CA.
12 Keenan Research Centre for Biomedical Science, St Michael's Hospital, Toronto, ON, Canada.
13 Department of Anesthesiology, Critical Care and Pain Medicine, Boston Children's Hospital, Boston, MA.
14 Center for Experimental and Molecular Medicine,Amsterdam University Medical Centers,Location Academic Medical Center, University of Amsterdam, Amsterdam, The Netherlands.
15 Department of Clinical Epidemiology, Biostatistics and Bioinformatics, Amsterdam University Medical Centers, Location Academic Medical Center, University of Amsterdam, Amsterdam, The Netherlands.
16 Guy's and St Thomas'NHS Foundation Trust, ICU support Offices, St Thomas'Hospital, London, United Kingdom. 17 School of Immunology and Microbial Sciences, KingsCollege London, London, United Kingdom.
18 Department of Emergency Medicine, Beth Israel Deaconess Medical Center and Harvard Medical School, Boston, MA.
19 Inflammatix, Burlingame, CA.
20 Department of Intensive Care Medicine, Nepean Hospital,Sydney, NSW, Australia.
21 Division of Pulmonary and Critical Care Medicine, Massachusetts General Hospital and Harvard Medical School, Boston, MA.
22 Department of Medicine, Center for Applied Genomics and Precision Medicine, Duke University School of Medicine, Durham,NC.
23 Division of Critical Care Medicine, Cincinnati Children's Hospital Medical Center and Cincinnati Children's Research Foundation, Cincinnati, OH.
24 Department of Critical Care Medicine, Peking University People's Hospital, Beijing, China.
The views expressed in this publication are those of the author(s)and not necessarily those of the NHS, the National Institute for Health Research or the Department of Health and social care. The funding source did not have any role in the design, conduct or interpretation of study results.
Dr. Seymour helped in designing the concept. Drs. DeMerle, Angus, and Seymour helped in designing. Drs. DeMerle, Baille, Brant, Calfee, Carcillo, Chang, Dickson, Evans, Gordon, Kennedy, Knight,Lindsell,Liu,Marshall,Randolph,Scicluna,Shankar-Hari, Shapiro, Sweeney, Talis, Tang, Thompson, Tsalik, van der Poll, van Vught, Wong, Yende, Zhao, and Seymour drafted the article and critically revised the article for important intellectual content.
Supplemental digital content is available for this article. Direct URL citations appear in the printed text and are provided in the HTML and PDF versions of this article on the journal's website (http://journals./ww.com/ccmjournal).
Dr. DeMerle's institution received funding from R35 GM119519-03 and T32HL007820. Dr. Calfee is supported in part by grants from the National Institutes of Health(NIH; HL140026).
Dr. Carcillo is supported in part by grants from the National Institutes of Health(Ro1GM108618). Dr. DeMerle is supported in part by grants from the National Institutes of Health(T32HL007820).Dr. Angus received funding from Ferring Pharmaceuticals, Bristol-Myers Squibb, Bayer AG, and Alung Technologies. Drs. Angus, Brant, Carcillo, Chang, Dickson, Kennedy, Lindsell, Liu, Randolph, Thompson, Tsalik, Wong, and Seymour received support for article research from the NIH.
Dr. Baillie received support for article re-search from Wellcome Trust/Charity Open Access Fund(COAF), and Research Councils UK. Dr. Calfee's institution received fund-ing from Roche/Genentech and Bayer, and she received funding from Roche/Genentech, Quark, CSL Behring, Bayer, Gen1e Life Sciences, and Vasomune. Drs. Carcillo's and Seymour's insti-tutions received funding from the National Institute of General Medical Sciences.
Drs. Chang's, Lindsell's, Liu's, Randolph's, Shapiro's, and Wong's institutions received funding from the NIH. Dr. Gordon's institution received funding from the National Institute for Health Research (NIHR) Research Professorship (RP-2015-06-18),NIHR Imperial Biomedical Research Centre, GlaxoSmithKline, and Bristo/Myers Squibb. Dr. Knight received support for article research from Wellcome Trust/COAF. Dr. Lindsell's institution received funding from the Centers for Disease Control and Prevention(CDC), Department of Defense, Marcus Foundation, Entegrion, Endpoint Health, and bioMerieux, and he disclosed he is a coinventor on patents related to risk stratification in septic shock. Dr. Marshall received funding from AM Pharma, AKPA Pharma, and the Society of Critical Care Medicine(Critical Care Medicine Associate Editor).
Dr. Randolph's institution re-ceived funding from the CDC, and she received funding from La Jolla Pharma. Dr. Shapiro's institution received funding from rapid pathogen screening, Baxter, and Inflammatix, and he received funding from Diagnostic Robotics. Dr. Sweeney received fund-ing from Inflammatix. Dr. Thompson's institution received funding from the National Heart, Lung, and Blood Institute, and he received funding from Bayer and Thetis. Dr. Tsalik disclosed that he is a founder and holds equity in Predigen; he receives salary support from the Durham VA Healthcare System and Duke University; and he has received salary support andlor grant funding (paid to his university) from the NIH, DARPA, DTRA, Karius, and Sanofi US. Dr. Wong disclosed that he and his institutions hold U.S.patents for sepsis biomarkers. Dr. Yende received funding from serving as consultant for expert testimony and he disclosed government work.
Dr. Knight is supported by a Wellcome Trust Investigator Award (204969/Z/16/Z) and the NIHR Oxford Biomedical Research Centre. Dr. Lindsell was supported in part by grants from the National Institutes of Health(R35GM126943,RO1HL149422), a research grant to VUMC from Endpoint Health, and is also listed as co-inventor on patents for endotyping and risk-stratification in pediatric septic shock. Dr. Liu is supported in part by grants from the National Institutes of Health(R35GM128672). Dr. Marshall is supported in part by grants from the Canadian Institutes of Health Research. Dr. Randolph is supported in part by grants from the National Institutes of Health(R21HD095228). Dr. Shankar-Hari is supported by the National Institute for Health Research Clinician Scientist Award(CS-2016-16-011).
Dr. Wong is supported in part by grants from the National Institutes of Health(R35 GM126943). Dr. Sweeney is an employee of, and shareholder in, Inflammatix. The remaining authors have disclosed that they do not have any potential conflicts of interest.
For information regarding this article, E-mail: demerlekm@upmc.edu
参考文献
1. Singer M, Deutschman CS, Seymour CW, et al: The third in-ternational consensus definitions for sepsis and septic shock (Sepsis-3).JAMA2016;315:801-810
2. Shankar-Hari M,Phillips GS,Levy ML,et al; Sepsis Definitions Task Force: Developing a new definition and assessing new clinical criteria for septic shock: For the third international consensus definitions for sepsis and septic shock(Sepsis-3). JAMA 2016;315:775-787
3. Rudd KE, Johnson SC, Agesa KM, et al: Global, regional,and national sepsis incidence and mortality, 1990-2017:Analysis for the global burden of disease study. Lancet 2020;395:200-211
4. Marshall JC: Why have clinical trials in sepsis failed? TrendsMol Med 2014;20:195-203
5. Seymour CW, Liu VX, Iwashyna TJ, et al: Assessment of clinical criteria for sepsis: For the third international consensus defi-nitions for sepsis and septic shock(Sepsis-3). JAMA 2016;315:762-774
6. Fohner AE, Greene JD, Lawson BL, et al: Assessing clinical heterogeneity in sepsis through treatment patterns and ma-chine learning. JAm Med Inform Assoc 2019;26:1466-1477
7. Reddy K, Sinha P, O'Kane CM, et al: Subphenotypes in crit-ical care: Translation into clinical practice. Lancet Respir Med 2020;8:631-643
8. Rhee C, Kadri SS, Danner RL, et al: Diagnosing sepsis is sub-jective and highly variable: A survey of intensivists using case vignettes. Crit Care 2016; 20:89
9. LevyMM,FinkMP,MarshallJC,etal;SCCM/ESICM/ACCP/ATS/SIS:2001 SCCM/ESICM/ACCP/ATS/SIS international sepsis definitions conference. Crit Care Med 2003; 31:1250-1256
10. Shankar-Hari M, Harrison DA, Rubenfeld GD, et al:Epidemiology of sepsis and septic shock in critical care units:Comparison between sepsis-2 and sepsis-3 populations using a national critical care database. Br J Anaesth 2017;119:626-636
11. Kaukonen KM, Bailey M, Pilcher D, et al: Systemic inflamma-tory response syndrome criteria in defining severe sepsis. N Eng/JMed 2015;372:1629-1638
12. Angus DC: Opening the debate on the new sepsis definition defining sepsis: A case of bounded rationality and fuzzy think-ing? Am J Respir Crit Care Med 2016;194:14-15
13. Schuler A, Wulf DA, Lu Y,et al: The impact of acute organ dys-function on long-term survival in sepsis. Crit Care Med 2018;46:843-849
14. Rhee C, Dantes R, Epstein L, et al: Incidence and trends of sepsis in US hospitals using clinical vs claims data, 2009-2014.JAM Med Assoc 2017;318:1241-1249
15. Stanski NL, Wong HR: Prognostic and predictive enrichment
in sepsis. Nat Rev Nephro/2020; 16:20-31
16. Opal SM, Laterre PF, Francois B, et al; ACCESS Study Group:Effect of eritoran, an antagonist of MD2-TLR4, on mortality in patients with severe sepsis: The ACCESS randomized trial. JAMA 20133091154-1162
17. Seymour CW, Gomez H, Chang CH, et al: Precision medicine for all? Challenges and opportunities for a precision medicine approach to critical illness.Crit Care 2017;21:257
18. Prescott HC, Calfee CS, Thompson BT, et al: Toward smarter lumping and smarter splitting: Rethinking strategies for sepsis and acute respiratory distress syndrome clinical trial design. Am J Respir Crit Care Med 2016;194:147-155
19. Seymour CW, Kennedy JN, Wang S, et al: Derivation, valida-tion, and potential treatment implications of novel clinical phe-notypes for sepsis. JAMA 2019; 321:2003-2017
20. Antcliffe DB, Burnham KL, Al-Beidh F, et al: Transcriptomic signatures in sepsis and a differential response to steroids. From the VANISH randomized trial. Am J Respir Crit Care Med 2019;199:980-986
21. Scicluna BP, van Vught LA, Zwinderman AH, et al; MARS Consortium: Classification of patients with sepsis according to blood genomic endotype: A prospective cohort study. Lancet Respir Med 2017;5:816-826
22. Bernard GR, Vincent JL, Laterre PF, et al; Recombinant Human Protein C Worldwide Evaluation in Severe Sepsis (PROWESS) Study Group: Efficacy and safety of recombinant human activated protein C for severe sepsis. N Eng/ J Med 2001;344:699-709
23. Yealy DM, Kellum JA, Huang DT, et al; ProCESS Investigators:A randomized trial of protocol-based care for early septic shock. NEng/JMed 2014;370:1683-1693
24. Maslove DM, Wong HR: Gene expression profiling in sepsis:Timing, tissue, and translational considerations. Trends Mol Med 2014;20:204-213
25. Maslove DM, Tang BM, McLean AS: Identification of sepsis subtypes in critically ill adults using gene expression profiling. Crit Care 2012;16:R183
26. Davenport EE, Burnham KL, Radhakrishnan J, et al: Genomic landscape of the individual host response and outcomes in sepsis:A prospective cohort study. Lancet RespirMed 2016; 4:259-271
27. Burnham KL, Davenport EE, Radhakrishnan J, et al: Shared and distinct aspects of the sepsis transcriptomic response to fecal peritonitis and pneumonia. Am J Respir Crit Care Med 2017;196:328-339
28. Xiao W, Mindrinos MN, Seok J, et al; Inflammation and Host Response to Injury Large-Scale Collaborative Research Program:A genomic storm in critically injured humans. J Exp Med 2011;208:2581-2590
29. Neyton LPA, Zheng X, Skouras C, et al: Multiomic defini-tion of generalizable endotypes in human acute pancre-atitis. bioRxiv. Preprint posted online May 11, 2019. doi:https://doi.org/10.1101/539569
30. Dickson RP: The microbiome and critical illness. Lancet Respir Med2016;4:59-72
31. Dickson RP, Singer BH, Newstead MW, et al: Enrichment of the lung microbiome with gut bacteria in sepsis and the acute respiratory distress syndrome. Nat Microbiol 2016;1:16113
32. Singer BH, Dickson RP, Denstaedt SJ, et al: Bacterial dissem-ination to the brain in sepsis. Am J Respir Crit Care Med 2018;197:747-756
33. Zaborin A, Smith D, Garfield K, et al: Membership and beha-vior of ultra-low-diversity pathogen communities present in the gut of humans during prolonged critical illness. mBio 2014;5:e01361-e01314
34. Lamarche D, Johnstone J, Zytaruk N, et al; PROSPECT Investigators; Canadian Critical Care Trials Group; Canadian Critical Care Translational Biology Group: Microbial dysbiosis and mortality during mechanical ventilation: A prospective ob-servational study.Respir Res 2018;19:245
35. Panzer AR, Lynch SV, Langelier C, et al: Lung microbiota is related to smoking status and to development of acute respi-ratory distress syndrome in critically ill trauma patients. Am J Respir Crit Care Med 2018;197:621-631
36. Taur Y,Jenq RR,Perales MA,et al:The effects of intestinal tract bacterial diversity on mortality following allogeneic hematopoi-etic stem cell transplantation. Blood 2014;124:1174-1182
37. Taur Y, Xavier JB, Lipuma L, et al: Intestinal domination and the risk of bacteremia in patients undergoing allogeneic hematopoi-etic stem cell transplantation. Clin Infect Dis 2012; 55:905-914
38. Kitsios GD, Morowitz MJ, Dickson RP, et al: Dysbiosis in the in-tensive care unit: Microbiome science coming to the bedside. J Crit Care 2017;38:84-91
39. Carcillo JA, Halstead ES, Hall MW, et al; Eunice Kennedy Shriver National Institute of Child Health and Human Development Collaborative Pediatric Critical Care Research Network Investigators: Three hypothetical inflammation pathobiology phenotypes and pediatric sepsis-induced mul-tiple organ failure outcome. Pediatr Crit Care Med 2017;18:513-523
40. Wong HR, Caldwell JT, Cvijanovich NZ, et al: Prospective clin-ical testing and experimental validation of the pediatric sepsis biomarker risk model. Sci Trans/Med 2019;11:eaax9000
41. Wong HR, Cvijanovich N, Allen GL, et al; Genomics of PediatricSIRS/Septic Shock Investigators: Genomic expression pro-filing across the pediatric systemic inflammatory response syndrome, sepsis, and septic shock spectrum. Crit Care Med 2009;37:1558-1566
42. Wong HR, Sweeney TE, Hart KW, et al: Pediatric sepsisendotypes among adults with sepsis. Crit Care Med 2017;45:e1289-e1291
43. Wong HR, Atkinson SJ, Cvijanovich NZ, et al: Combining prog-nostic and predictive enrichment strategies to identify children with septic shock responsive to corticosteroids. Crit Care Med 2016;44:e1000-e1003
44. Wong HR, Cvijanovich NZ, Anas N, et al: Endotype transi-tions during the acute phase of pediatric septic shock reflect changing risk and treatment response. Crit Care Med 2018;46:e242-e249
45. Sweeney TE, Azad TD, Donato M, et al: Unsupervised anal-ysis of transcriptomics in bacterial sepsis across multiple datasets reveals three robust clusters. Crit Care Med 2018;46:915-925
46. Ruppel H, Bhardwaj A, Manickam RN, et al: Assessment of electronic health record search patterns and practices by practitioners in a large integrated health care system. JAMA Netw Open 2020;3:e200512
47. Komorowski M, Celi LA, Badawi O, et al: The artificial intelli-gence clinician leams optimal treatment strategies for sepsis in intensive care. Nat Med 2018; 24:1716-1720
48. Angus DC: Optimizing the trade-off between learning and doing in a pandemic. JAMA 2020; 323:1895-1896
专家述评
赵慧颖 北京大学人民医院重症医学科
马朋林 贵黔国际总医院重症医学科
脓毒症的定义为感染引起失调的宿主反应所导致的致命性器官功能不全,是ICU的主要疾病之一,具有非常高的发病率和病死率。但近年来大量关于脓毒症治疗的RCT阴性结果提示脓毒症是一个存在高度异质性的临床综合征,即具有广泛的临床或生物学特征、且对治疗反应各异。因此,迫切需要对脓毒症进行分型以进一步探索更加符合病人需要的个体化治疗对策。然而,随着脓毒症分型相关的研究越来越多,一系列问题亦相应出现,如亚型的定义、分型的目的、所采用的分型方法、相关研究结果的意义以及如何知道所分之亚型是否真实存在等。该文旨在帮助认识与理解读脓毒症亚型、提供不同方法学所研发的脓毒症亚型的解读原则、并指导临床医师在工作中如何考量脓毒症亚型问题。
首先,作者综述了脓毒症分型的一些基本问题。在回答为何脓毒症分型存在困难这一问题时指出
(1)脓毒症的诊断本身就存在一定争议,尤其合并基础器官功能不全的患者。并且应用聚类分析进行分型,聚类边缘的个体分型并不明晰;
(2)统计学方法学并未统一,尚未有最佳方案处理缺失数据;(3)脓毒症亚型研究的数据来源包括队列、RCT、临床大数据等,哪种更佳,以及是否需要多种数据进行联合,目前还不清楚。
这些问题是当前开展脓毒症分型的主要障碍。
在分析脓毒症分型目的时强调
(1)将患者分型,判断预后;
(2)探索不同亚型的生物学机制;
(3)对不同的亚型采取精准治疗,进而改善患者预后。
同时作者提出,当前采用的脓毒症亚型的用词也存在问题,如亚型(subgroup, subset, subclass, subtype)、表型(phenotype,来自临床大数据)、亚表型(subphenotype,来自临床数据)、内型(endotype,多来自基因组数据)等,目前还不统一,容易引起混淆。
其次,在研究方法学上,作者提出脓毒症亚型研究的数据来源包括既往的RCT和观察性队列研究,RCT的优势是数据全面并能分析治疗效果,缺点是样本量小,纳入病例不能反映真实世界;观察性队列研究来自电子病历大数据,样本量大,变量丰富,
但多存在缺失数据,不宜判断治疗效果。作者以往的ENECA研究的经验是将这两种数据结合进行研究,相互弥补不足。作者还提出数据变量选择上目前主要包括RNA转录基因组学和临床数据等。基因组学数据在判断预后、区分治疗效果的同时,还能探索不同亚型的机制。基因组学数据亟待解决问题为时效性、费用、可重复性、技术需简单化等。随着科技的发展,期待将分子生物学的亚型应用于床旁,并和临床数据结合。
在统计学方法选择方面作者提出统计学目的包括
(1)具有相似的变量;
(2)根据不同或相似的结果;
(3)根据不同的治疗反应性。
如果仅是将相似特征的病人分为同一类型,选用的是非监督聚类方法;如果需要基于预后或治疗反应性分型,则采用监督模型,方法应为目的服务,合理且可解释。
最后,作者对脓毒症分型的意义提出以下期许:
(1)脓毒症亚型应具有生物学合理性、预测治疗反应性的能力、在其他数据中的一致性和可重复性;
(2)将临床、生物学、分子学的数据结合;
(3)选取固定变量,应用深度机器学习方法,建立成熟的床旁模型,实现脓毒症床旁分型和治疗指导。
总之,脓毒症的分型目前的研究仍处于初级阶段,有很多问题亟待解决,我们期待能建立可靠、可重复、床旁易获取的脓毒症分型模型,并能广泛推广,进而做到真正的精准治疗!
艾宇航 中南大学湘雅医院
脓毒症是一种广泛异质的综合征,最近被定义为感染引起失调的宿主反应所导致的致命性器官功能障碍,是重症医学最常面临的挑战。然而,自脓毒症早期复苏目标实施以来,支持性的监测治疗和新疗法的试验一直令人失望,脓毒症的病死率没有明显减少,这促使临床医生及研究者探索脓毒症的亚型以及针对亚型的精准治疗是否能够改善患者预后。
疾病的分型依据不同会导致很多的分型结果,不同的分型可能会对应不同的治疗和预后;不同亚型之间可能存在交叉,不同研究者的分型方法、不同研究结论也无可比性,这些问题作者一一提出并进行讨论:
1) 脓毒症亚型的语义基础,
2) 亚型的概念目标,
3) 关于研究设计、数据源和统计方法的考虑,
4) 新兴数据类型的作用,
5)如何判断亚型是否代表其“真实性”。并为广泛的脓毒症亚型研究提出一个框架。
目前尚无广泛接受的脓毒症亚型,作者所提出的框架可能为进一步的脓毒症亚型的研究提供方法以及应具备的细节,对于临床医生而言从亚型的机制入手应该能更好地针对某一亚型的脓毒症病人提供精准的医疗服务,从而有可能改善患者预后。
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言