Nature子刊:再取进展!西湖大学王程程/占谢超揭示酵母组蛋白去乙酰酶复合体Rpd3L的组装机制
2023-04-23 iNature iNature 发表于上海
该研究展示了出芽酵母SIN3-HDAC复合物Rpd3L的冷冻电镜结构,平均分辨率为2.6 Å。该结构揭示了两个不同的臂(ARM1和ARM2)悬挂在由两个线圈结构域形成的t形支架上。
SIN3-HDAC(组蛋白去乙酰化酶)复合物在促进局部组蛋白去乙酰化以调节染色质可及性和基因表达方面具有重要作用。但人们对SIN3-HDAC复合体内亚基的排列以及组织机制知之甚少
2023年4月20日,西湖大学王程程和占谢超共同通讯在Nature Structural & Molecular Biology在线发表了题为“Structure of a SIN3–HDAC complex from budding yeast”的研究论文,该研究展示了出芽酵母SIN3-HDAC复合物Rpd3L的冷冻电镜结构,平均分辨率为2.6 Å。该结构揭示了两个不同的臂(ARM1和ARM2)悬挂在由两个线圈结构域形成的t形支架上。
在每只手臂中,Sin3与不同的亚基相互作用,为组蛋白去乙酰化酶Rpd3创造不同的环境。ARM1处于抑制状态,Rpd3的活性位点被阻断,而ARM2处于开放构象,Rpd3的活性位点暴露于外部空间。研究观察到的Rpd3L的不对称结构与其他一类HDAC配合物的结构不同。该研究揭示了SIN3-HDAC复合物的组织机制,并提供了它将组蛋白去乙酰化酶靶向染色质的相互作用模式的见解。
另外,2023年4月19日,西湖大学占谢超及王程程共同通讯在Cell Discovery(IF=38)在线发表题为“Two assembly modes for SIN3 histone deacetylase complexes”的研究论文,该研究揭示了SIN3组蛋白去乙酰化酶复合物的两种组装模式。在SIN3L的结构中,每个Sin3亚型(Pst1和Pst3)与一个组蛋白去乙酰化酶Clr6和一个含WD40的蛋白Prw1相互作用,形成两个裂片。这两个叶分别由来自Sds3/Dep1和Rxt2/ Png2的两个垂直线圈结构域桥接。在SIN3S的结构中,只有一个叶瓣由另一个Sin3异构体Pst2组织;Cph1和Cph2分别与Eaf3分子结合,为组蛋白识别和结合提供了两个模块。值得注意的是,SIN3L中的Pst1叶瓣和SIN3S中的Pst2叶瓣具有相似的构象,它们的去乙酰化酶活性位点暴露在空间中;然而,SIN3L中的Pst3 Lobe处于致密状态,其活性中心埋在内部并被阻断。总之,该研究工作揭示了SIN3/HDAC复合物实现特异性靶向的两种经典组织机制,并为研究组蛋白去乙酰化酶复合物提供了一个框架(点击阅读)。

组蛋白去乙酰化在基因表达、表观遗传和染色质结构调控中具有重要作用。HADCs最早是在20世纪90年代发现的。先前的研究表明,锌依赖性I类HADCs的催化结构域在氨基酸序列和三维(3D)结构水平上是相似的,导致底物选择性有限。事实上,I类HADCs可作为多种转录调控复合物的催化亚基,包括SIN3-HDAC、NuRD、CoREST、SMRT-NCoR和MiDAC。越来越多的人认识到,这些复合物对于HADCs通过多种机制靶向染色质以实现高度特异性调控至关重要。SIN3-HDAC复合物,从酵母到人类都是保守的,与许多细胞过程有关,包括发育、生长、分化和衰老。此外,它们还为多种疾病提供了重要的治疗靶点,包括三阴性乳腺癌和胰腺癌。
在哺乳动物中,Sin3有Sin3A和Sin3B两种同工异构体,分别形成两种不同的复合物。人类Sin3A和Sin3B的杂合突变或缺失已被确定为导致颅面缺陷综合征性智力残疾的遗传因素。出芽酵母Saccharomyces cerevisiae只有一个Sin3的异构体,它与HDAC同源物Rpd3形成大的和小的复合物,分别称为Rpd3L和Rpd3S。Rpd3L复合物使启动子区域的组蛋白去乙酰化,而Rpd3S复合物靶向转录区域抑制基因内转录起始。先前的研究表明,一些组分(如Sin3、Sds3、Dep1、Rxt2和Pho23)的突变导致与Rpd3突变体相似的表型,表明它们是该复合物在体内正常功能所必需的。
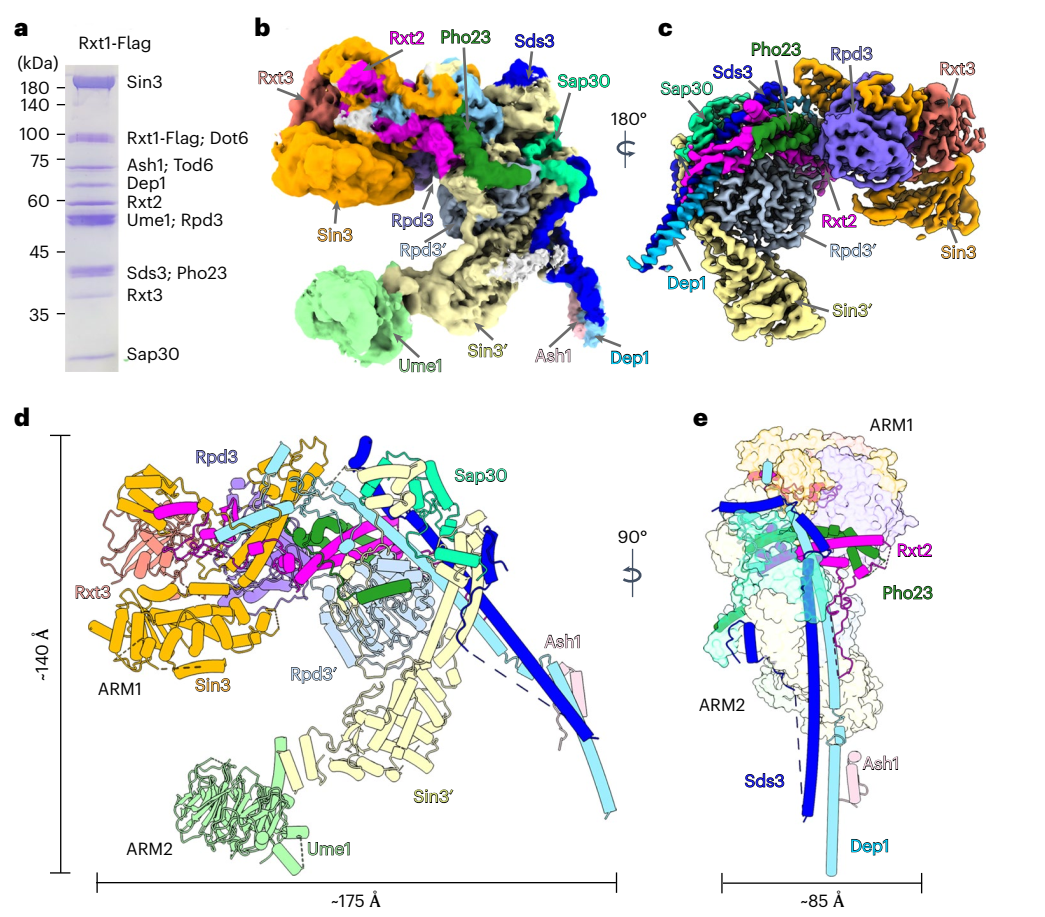
Rpd3L复合物的冷冻电镜结构(图源自Nature Structural & Molecular Biology )
尽管它在染色质修饰中起着关键作用,但人们对SIN3-HDAC复合体内亚基的排列知之甚少。完整的SIN3-HDAC复合物的复杂和灵活的性质可能阻碍了其结构研究。该研究报告了S. cerevisiae Rpd3L配合物的冷冻电镜结构,平均分辨率为2.6 Å。该研究确定了S. cerevisiae Rpd3L复合物的结构,揭示了SIN3-HDAC复合物独特而复杂的结构。
Sds3-Dep1和Pho23-Rxt2提供了两个螺旋结构域,形成了一个t形支架,ARM1和ARM2被挂在上面,而Sin3与不同的亚基相互作用,在每个臂上为组蛋白去乙酰化酶Rpd3创造了不同的环境。ARM1中Rpd3的活性位点被Rxt2_loop1阻断,而ARM2中Rpd3 的活性位点暴露于外部空间。值得注意的是,Rpd3L的不对称结构与其他一类HDAC配合物的现有结构有很大不同。该研究结果提供了对SIN3-HDAC复合物组织的深入了解,以及HDAC如何与Sin3结合以使去乙酰化酶活性的机制理解。
西湖大学生命科学学院施一公团队副研究员王程程、占谢超为本文的共同通讯作者,西湖大学博士生郭舟妍和储晨为共同第一作者。施一公教授为本研究提供了大力支持和建议。西湖大学博士生卢怡辰、原西湖大学助理研究员张晓峰、西湖大学理学院研究员吴明轩和博士生肖义杭参与了本研究的部分工作。北京协和医院黄超兰教授和高帅鑫高级工程师参与了质谱部分的工作。
本研究得到了西湖大学冷冻电镜平台、高性能计算中心和质谱与代谢组学平台的大力支持。本研究获得了国家自然科学基金委、西湖大学、西湖教育基金会的相关经费支持。
原文链接:
https://www.nature.com/articles/s41594-023-00975-z
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言








收获很多
41