刘颖团队利用CRISPR筛选,鉴定mTORC1感应氨基酸的调控蛋白ILF3
2023-04-11 “生物世界”公众号 “生物世界”公众号 发表于上海
该研究通过全基因组CRISPR筛选鉴定了mTORC1感应氨基酸过程中的调控蛋白,并解析了其中一个新的调控蛋白ILF3调控mTORC1的分子机制,为mTORC1感知氨基酸的机制研究提供了支持。
mTORC1信号通路是通过mTORC1的激酶活性以磷酸化不同底物进而调控细胞代谢的核心通路之一。氨基酸等营养物质通过一系列信号转导过程将其浓度信号传递给mTORC1。不同的氨基酸通过直接结合相应的感知蛋白,影响感知蛋白与GATOR复合体(包括亚复合体GATOR1和GATOR2)的结合,进而控制Rag GTPase的GTP负载以调节mTORC1的溶酶体定位。但在此信号传递过程中,仍旧有一些问题尚待解答,意味着可能还有一些调控蛋白尚未得到鉴定。
2023年4月10日,北京大学未来技术学院、北大-清华生命科学联合中心刘颖课题组在 Nature Cell Biology 期刊发表了题为:Genome-wide CRISPR screens identify ILF3 as a mediator of mTORC1-dependent amino acid sensing 的研究论文。
该研究通过全基因组CRISPR筛选鉴定了mTORC1感应氨基酸过程中的调控蛋白,并解析了其中一个新的调控蛋白ILF3调控mTORC1的分子机制,为mTORC1感知氨基酸的机制研究提供了支持。

在此项研究中,为了在全基因组范围内寻找潜在的mTORC1调控蛋白,作者利用了数年前由张锋课题组开发的CRISPR全基因组敲除慢病毒筛选系统。进行筛选的重要前提之一是需要找到一个理想的readout,其既能够特异性且敏感地监测mTORC1活性,又能与基于FACS的细胞分选兼容。不幸的是,一些在免疫印迹实验中常用来指征mTORC1活性的mTORC1直接底物,如S6K1、4EBP1、TFEB和ULK1,未能较好地满足第二个要求。因此,研究人员将注意力放在了一个mTORC1-S6K1磷酸化级联反应的效应蛋白,即核糖体蛋白S6上。结合前人的实验结果,作者通过免疫印迹和免疫荧光实验验证了S6的S235/6的磷酸化(pS6)信号对氨基酸饥饿和再刺激十分敏感。最重要的是,FACS实验确认了pS6荧光强度在氨基酸饥饿的细胞群与氨基酸再刺激细胞群之间能够得到良好的区分。提示pS6能作为理想的全基因组CRISPR筛选readout。
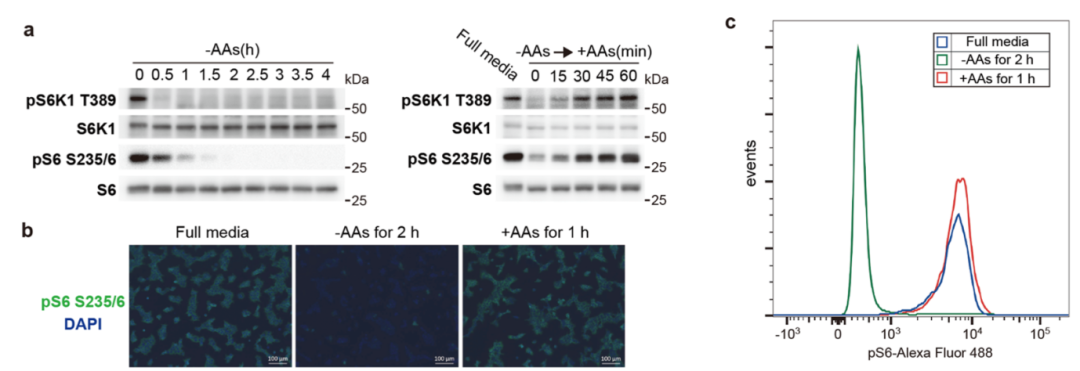
图1. 全基因组CRISPR筛选mTORC1调控蛋白的readout。(a)pS6K1和pS6均能在免疫印迹实验中显示出对氨基酸浓度变化的敏感性;(b)pS6信号在免疫荧光实验中呈现对氨基酸饥饿和再刺激的敏感性;(C)FACS实验证明pS6荧光强度在氨基酸饥饿与再刺激的细胞群之间能够得到良好的区分
找到了合适的筛选readout后,作者在氨基酸饥饿(-AAs)或饥饿后再刺激(+AAs)条件下进行基于FACS的全基因组CRISPR筛选。筛选结果成功富集到了mTORC1感应氨基酸过程中多个已知的关键基因,例如GATOR1复合体的所有3个组分,提示了筛选结果的可靠性。为了寻找新的mTORC1调控蛋白,通过进一步筛选,作者后续将重点放在了一个之前未被报道能影响mTORC1活性的基因ILF3上,并通过一系列实验证明ILF3的一个isoform,即ILF3i1,在mTORC1感知氨基酸过程中发挥着负调控作用,且这种调控不像氨基酸感知蛋白一样对氨基酸有选择性。
Rag GTPase在感知氨基酸信号后直接募集mTORC1到溶酶体表面。为了验证ILF3是否也是通过此路径调控mTORC1,作者首先通过基因敲除及溶酶体亲和纯化手段证明了ILF3在Rag GTPase的上游发挥着调控mTORC1溶酶体移位的功能。后续ILF3与Rag GTPase上游的多个已知关键节点的上位分析实验显示ILF3在GATOR复合体的下游调控mTORC1。为了进一步探索ILF3调控mTORC1的分子机制,作者用免疫共沉淀检测了ILF3是否可能与mTORC1感知氨基酸过程中的关键调控蛋白或蛋白复合体之间有相互作用。
由此,作者发现,ILF3能以一种氨基酸不敏感的方式结合GATOR复合体。进一步的研究发现ILF3本身有部分定位于溶酶体,并且能将GATOR复合体靶定在溶酶体表面,由此阐释了ILF3的对mTORC1信号的调控机制。

图2. ILF3调控mTORC1信号通路的机制
ILF3在线虫至人均有其同源基因。那其在调控mTORC1上的功能是否也是物种保守的呢?自噬活性及TFEB的出入核情况是mTORC1下游的另外2个通用的readout,并且在人、小鼠及线虫上均有成熟且灵敏的荧光融合蛋白报告系统以供检测。通过利用此系统,研究人员证明了从线虫到小鼠细胞再到人源细胞,ILF3都能保守地调控mTORC1的活性。mTORC1信号在多个物种中对机体的衰老具有显着地调节作用,最后研究者也利用线虫模型证明了ILF3能通过影响mTORC1的活性调节线虫的衰老进程。
综上,该通过全基因组CRISPR敲除筛选方式鉴定了一个mTORC1感知氨基酸过程中新的调控基因ILF3,解析了ILF3调控mTORC1的分子机制,并明确了ILF3在机体衰老过程中的作用。
北京大学未来技术学院博士后严国楷为论文第一作者,刘颖教授为论文通讯作者,北京大学博士研究生杨金鑫、李雯、郭澳、关嘉良均为本项工作做出重要贡献。
原始出处:
Yan, G., Yang, J., Li, W. et al. Genome-wide CRISPR screens identify ILF3 as a mediator of mTORC1-dependent amino acid sensing. Nat Cell Biol (2023). https://doi.org/10.1038/s41556-023-01123-x.
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言