引用本文
陈瑛瑶,储香玲,俞 昕,等. 免疫检查点抑制剂疗效相关预测模型的研究进展[J]. 中国癌症杂志, 2023, 33(1): 61-70.
第一作者:陈瑛瑶(ORCID: 0000-0002-2804-0851),博士在读。
通信作者:苏春霞(ORCID: 0000-0003-1632-9487),博士,主任医师、教授。
免疫检查点抑制剂疗效相关预测模型的研究进展
陈瑛瑶,储香玲,俞 昕,苏春霞
同济大学附属上海市肺科医院肿瘤科,上海 200433
[摘要] 免疫检查点抑制剂(immune checkpoint inhibitor,ICI)的应用让肿瘤治疗取得了新突破,但不同患者接受免疫治疗后疗效差异较大,仅部分患者能够从中获益。通过检测一些生物标志物可以预测ICI的疗效,如程序性死亡[蛋白]配体-1(programmed death ligand-1,PD-L1)及肿瘤突变负荷(tumor mutation burden,TMB)等。除此之外,目前已有多项研究基于肿瘤患者的基因组学、转录组学或影像组学等数据,筛选多个生物标志物并建立免疫治疗效果相关预测模型。这类模型具备严谨的建立及验证流程,能够纳入更多肿瘤免疫相关变量,有助于提高对ICI疗效的预测能力。本文就肿瘤免疫治疗效果相关预测模型进行综述,以期为免疫治疗获益人群的筛选提供新思路。
[关键词] 免疫治疗;疗效;预后;预测模型
[Abstract] The use of immune checkpoint inhibitor (ICI) has revolutionized the treatment among patients with various types of tumors. However, only some patients can benefit from ICI. The identification of predictive markers of response to treatment in patients is required, such as programmed death ligand-1 (PD-L1) and tumor mutation burden (TMB). Besides, there have been numerous studies using sequence and radiomics data based on large populations to explore the factors related to the efficacy, and to establish the prediction model. This kind of model has a rigorous establishment and validation process, can include more tumor immune related variables, and is helpful to improve the prediction ability of the efficacy of ICI. This paper reviewed the establishment of immunotherapy prediction models and provided new thoughts pertaining to screening the potential beneficiaries from immunotherapy.
[Key words] Immunotherapy; Efficacy; Prognosis; Prediction model
免疫检查点抑制剂(immune checkpoint inhibitor,ICI)旨在激活肿瘤免疫循环,通过阻断肿瘤免疫逃逸,利用自身免疫细胞,特别是T细胞来消灭肿瘤细胞[1]。以程序性死亡[蛋白]-1(programmed death-1,PD-1)或程序性死亡[蛋白]配体-1(programmed death ligand-1, PD-L1)抑制剂、细胞毒性T淋巴细胞相关抗原4(cytotoxic T lymphocyte associated antigen-4, CTLA-4)抑制剂为代表的ICI已被广泛应用于临床,显著延长了肿瘤患者的生存时间[2-3]。以晚期黑色素瘤为例,使用帕博利珠单抗治疗的患者5年生存率已提升至38.7%[4-6]。然而,并非所有患者都能从免疫治疗中获益,在大多数晚期实体瘤患者中,ICI单药治疗的有效率约为23%[6]。此外,初次免疫治疗有效的患者也可能在后续治疗中产生耐药性[7-8]。因此,建立有效的免疫治疗效果相关预测模型十分重要。
目前,已获美国食品药品管理局(Food and Drug Administration,FDA)批准的免疫治疗生物标志物包括PD-L1、肿瘤突变负荷(tumor mutation burden,TMB)及微卫星不稳定性/错配修复缺陷(microsatellite instability/mismatch repair deficiency,MSI/dMMR)[9]。研究较多的生物标志物还包括特定的基因突变、肿瘤浸润免疫细胞、循环肿瘤DNA(circulating tumor DNA,ctDNA)及肠道微生物群等[10-12]。然而,标志物的预测效能往往会受到肿瘤类型、样本量及检测方法等因素的影响[13-14]。为提高生物标志物的有效性和普适性,有必要探索出更加客观、可行的疗效预测工具。
预测模型是通过统计学方法对不同途径获取的不同类型数据进行整合,并从中计算出个别患者发生特定临床事件的概率,进而对患者治疗获益进行评估的一种量化工具[15]。预测模型建立的主要步骤包括数据收集、变量筛选、模型构建、验证及评估[16]。目前在肿瘤领域,预测模型已尝试应用于多种临床场景,并取得了较大进展。例如,利用遗传学特征、人口学特征、肺功能检查结果等数据建立肺癌患病风险预测模型,该类模型能够优化肺癌筛查策略,从而实现癌症的早筛、早诊[17]。同样,根据相关实验室检验数据等建立的晚期肝癌患者生存预测模型能够精准地预测肝癌患者的短期死亡风险,进而辅助医师调整患者的后续治疗方案[18]。近年来,随着个体多变量预后预测模型透明化报道声明[15]的发布,预后预测模型的建立逐渐公开化、系统化,形成了一套完整的模型评估标准。本文通过汇总基于不同数据源和不同方法所建立的免疫治疗效果相关预测模型(表1),并对现有的预测模型进行梳理和评价,以期为免疫治疗获益人群筛选途径的探索提供新思路。
1
统计学模型
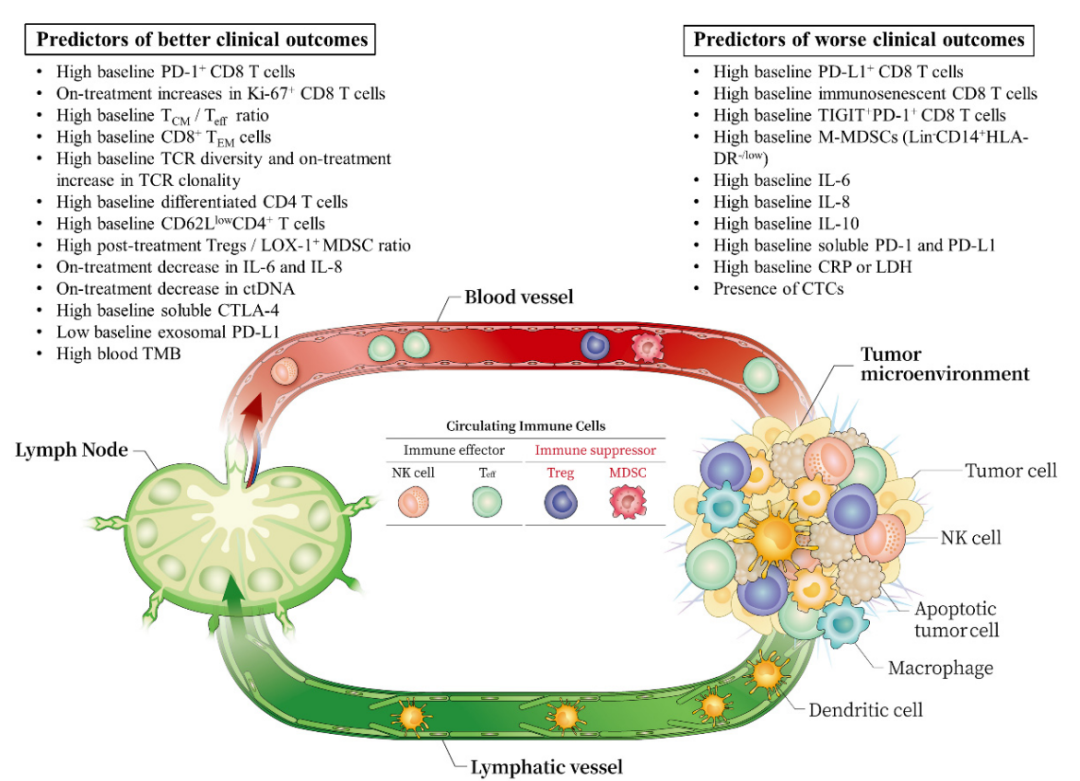
随着测序技术的不断发展与普及,肿瘤研究进入了多维度、多组学的大数据时代。一方面,肿瘤基因组学、转录组学、蛋白质组学等数据能够让研究者在分子层面对肿瘤的发生、发展机制及治疗模式进行探索[45-46];另一方面,癌症基因组图谱(The Cancer Genome Atlas,TCGA)和基因表达汇编(Gene Expression Omnibus,GEO)等公共数据库的建立,进一步开拓了研究人员获取有效数据的途径。基于肿瘤多组学数据,我们可以采用统计学算法筛选出有意义的自变量并建立临床预测统计学模型。此外,机器学习算法的引入能够帮助我们实现大数据的高效处理。以深度学习为例,利用卷积神经网络输出计算机体层成像(computed tomography,CT)图像或组织病理学图像的特征,结合影像组学分类器,可以建立基于医学图像的肿瘤免疫治疗效果预测模型[47](图1)。
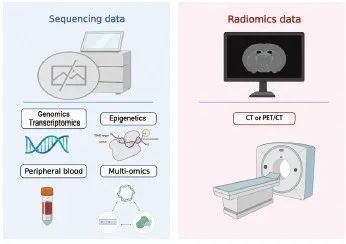
图1 ICI疗效预预测统计学模型概况
Fig. 1 Overview of statistical models for predicting the efficacy of ICI
1.1 基于测序数据构建模型:单一组学或多组学联用
研究[48]显示,癌症患者原发性耐药的发生率为7%~27%,继发性耐药的发生率高达20%~44%。其中遗传信息变异可能会导致肿瘤抗原免疫原性不足、T细胞耗竭、肿瘤微环境抑制等,进而导致耐药[49]。因此从不同的数据维度挖掘患者的遗传信息,是建立疗效预测模型的思路之一。
1.1.1 基于基因组学及转录组学相关数据
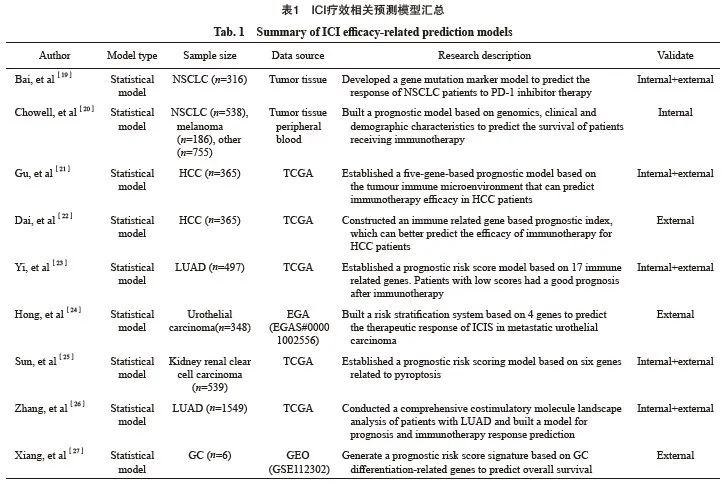
Bai等[19]提取316例接受免疫治疗的非小细胞肺癌(non-small cell lung cancer,NSCLC)患者的DNA测序数据,筛选并建立一个由8种基因(TP53、KRAS、STK11、EGFR、PTPRD、KMT2C、SMAD4和HGF)组成的基因突变标记(genomic mutation signature,GMS)风险评分模型,并根据GMS评分将患者分为高GMS(评分>0.565)组和低GMS(评分≤0.565)组。结果显示,与低GMS组患者相比,高GMS组患者有更长的无进展生存期(progression-free survival,PFS)[6.63个月 vs 2.50个月,风险比(hazard ratio,HR)=0.41,P<0.000 1]及总生存期(overall survival,OS)(未达到 vs 13.00个月,HR=0.53,P=0.027 5),这一发现在内部验证队列及外部验证队列中均得到证实,作者进一步分析发现,GMS评分与PD-L1检测联合能进一步提高对PD-1抑制剂疗效的预测能力,有较大的潜力成为临床实用的辅助预测工具。
为了更全面地评估肿瘤基因组学特征, Chowell等[20]在研究中纳入16种不同癌症类型的患者(n=1 479),包括NSCLC、黑色素瘤、肝细胞癌、乳腺癌等,整合了TMB、MSI、拷贝数变异分数(fraction of copy number alteration,FCNA)等多种基因组学数据,并结合临床特征及人口统计学数据,建立了免疫治疗临床反应预测模型[曲线下面积(area under curve,AUC)>0.80]。该模型在不同癌症类型中均表现良好,有较高的普适性。
除探讨肿瘤的基因组学特点外,对其基因表达特征进行分析也是很多研究的聚焦点。Dai等[21]、Gu等[22]和Yi等[23]均利用TCGA数据库,提取肿瘤免疫相关基因表达特征,据此构建免疫治疗效果预测模型。Hong等[24]鉴定了肿瘤缺氧相关基因,建立了一个基于4种基因(TKTL1、JMJD6、IRS2、ANXA2)的风险分层系统,可以通过评估肿瘤的缺氧微环境来预测转移性尿路上皮癌患者的治疗反应(AUC=0.71)。另有研究[25]筛选出6种细胞焦亡相关基因并建立风险评分系统,证实不同风险人群的肿瘤免疫细胞浸润有较大差异,高风险评分的患者对ICI有较好的应答(AUC=0.728)。基于mRNA表达,而非基因突变或表观遗传修饰来预测疗效不需要对患者进行全基因组测序,临床可行性高。但上述模型的数据维度都较为单一,且对于所筛选出来的基因如何影响ICI疗效的理论机制有待进一步实验验证。
Zhang等[26]对肺腺癌患者中来自B7-CD28和肿瘤坏死因子(tumor necrosis factor,TNF)家族的共刺激分子进行研究,初步构建了一个基于5种基因(CD40LG、TNFRSF6B、TNFSF13、 TNFRSF13C和TNFRSF19)的共刺激分子表达签名(costimulatory molecule-based signature,CMS),用以建立风险评分系统,结果表明,高风险组的OS显著低于低风险组(HR=2.043 5,P <0.000 1)。该研究率先对肺腺癌患者进行最全面的共刺激分子图谱分析,有助于进一步优化癌症的免疫治疗策略。
近年来,单细胞测序技术不断普及,单细胞转录组测序(single cell RNA-sequence,scRNA-seq)可以鉴别不同的细胞类型与其表达的基因,从单细胞层面更加精确地揭示肿瘤内与肿瘤间的异质性。Xiang等[27]收集胃癌患者肿瘤组织样本的scRNA-seq数据,根据胃癌细胞分化轨迹分析确定了3种不同分化状态的细胞亚群,其中亚群Ⅰ/Ⅱ与代谢紊乱有关,亚群Ⅱ与缺氧耐受有关,亚群Ⅲ与免疫相关通路有关。由此构建了基于胃癌细胞分化相关基因(gastric cancer differentiation-related gene,GDRG)的风险评分模型来预测患者接受免疫治疗后的生存情况。此外,作者结合风险评分、年龄、TNM分期和N分期这四种预后因素构建列线图预后模型,为预测患者的OS提供了一种可视化的方法。
在一项针对NSCLC的单细胞转录组图谱研究中,Leader等[28]聚焦肿瘤免疫微环境特征,探讨免疫细胞表型的变化,该研究使用scRNA-seq、转录组与细胞表位测序、T细胞受体测序分析了35例早期NSCLC病变中的361 929个细胞,构建了迄今为止最大的早期肺癌免疫反应scRNA图谱。研究发现,激活型T细胞、免疫球蛋白G(immunoglobulin G,IgG)+浆细胞及MoMΦ-Ⅱ细胞等与肺癌的发生有很高的相关性,作者将其称为肺癌激活模块(lung cancer activation module,LCAM),并建立了LCAM评分模型。根据该评分模型将患者分层,高LCAM评分(LCAM前25%)的患者能从免疫治疗中获得更长的PFS(7.3个月 vs 2.7个月,HR=0.58),提示LCAM可能是反映患者免疫激活特征的一个可靠指标。
1.1.2 基于表观遗传学相关数据
除分析肿瘤基因组及转录组数据外,表观遗传学调控也在肿瘤免疫反应过程中发挥重要作用。表观遗传是指基于非基因序列改变所致基因表达水平的变化,包括DNA甲基化、组蛋白修饰、染色体重塑和非编码RNA调控等[50]。表观遗传学调控在肿瘤免疫反应过程中发挥着重要作用。Lu等[29]从TCGA数据库中获取子宫内膜癌患者的TMB及miRNA表达相关数据,将患者分为高TMB组和低TMB组,并筛选出两组间差异表达的miRNA,构建基于miRNA的风险评分模型。该评分能够精确地反映患者的TMB水平,并预测其对ICI的治疗反应(AUC=0.911)。此模型在TMB的基础上筛选表观遗传学变量,有很高的预测精准度,但缺乏对这些免疫相关的miRNA如何调控肿瘤免疫活性的进一步研究。
肿瘤免疫微环境中免疫细胞的表达调控,特别是长链非编码RNA(long non-coding RNA,lncRNA)介导的免疫细胞浸润,也影响着免疫治疗患者的预后。LncRNA已被证明可通过多种途径调节细胞自噬,从而影响肿瘤的发生、发展。近10年来,已有研究[51]将自噬相关lncRNA作为免疫治疗生物标志物进行了深入探索,并证明其与癌细胞的生长、生存、染色质修饰、基因组印迹等重要生物学行为密切相关。Gao等[30]从TCGA数据库中提取375个胃癌组织和32个正常组织的转录组数据并进行分析,确定了与自噬相关的lncRNA,并结合临床数据,进一步筛选出具有预后价值的lncRNA,构建lncRNA风险评分预后预测模型(AUC=0.713)。风险评分与免疫治疗患者的OS率呈负相关,与高风险患者相比,低风险患者的M0巨噬细胞、活化CD4+ T细胞、肥大细胞等浸润均增加。该模型预测能力良好,但仍需要在样本量更大的外部队列中进行临床验证。
1.1.3 基于外周血样本相关数据
肿瘤患者的外周血中可能存在ctDNA、循环肿瘤细胞(circulating tumor cell,CTC)、肿瘤标志性蛋白等物质,这些指标能够反映肿瘤的发生、发展状态[52]。目前,利用外周血样本进行预后预测的方法也已逐渐成熟。Nabet等[31]根据接受免疫治疗的NSCLC患者(n=273)的PD-L1表达水平、循环CD8+ T细胞及血液TMB开发并验证了免疫治疗效果动态评估模型(durable immunotherapy response estimation by immune profiling and ctDNA-pre-treatment,DIREct-Pre),高DIREct-Pre评分的患者较低评分患者有更长的PFS(HR=2.66,P=0.012)。此外,作者引入早期治疗时的ctDNA水平,并在DIREct-Pre的基础上对模型进行了改进,构建了基于治疗前及早期治疗时的ctDNA水平和外周血CD8+ T细胞水平的评估模型(durable immunotherapy response estimation by immune profiling and ctDNA-on-treatment,DIREct-On),并证明了DIREct-On明显优于单独应用量化ctDNA进行预测[净重新分类指数(net reclassification index,NRI)=-1.13,P<0.01]。
在另一项基于外周血样本的研究[32]中,作者收集多种癌症类型患者(n=120)基线及第1次治疗评估前的外周血样本,提取外周血单个核细胞(peripheral blood mononuclear cell,PBMC)进行流式细胞免疫表型分析,建立了外周血免疫细胞标签(peripheral blood immune cells-based signature,BICS)。采用surv-cutpoint函数将患者进行分组,高BICS组患者对ICI治疗反应较差,OS更短(HR=4.21,P<0.001)。此外,作者还构建了基于外周血免疫细胞标志物的机器学习SVM-RFE分类器模型来预测患者的临床获益,该模型的预测效能在各个癌症类型中表现稳定。
上述两个模型避免了对肿瘤组织取样的有创操作,易于实现动态监测,后续应进一步在样本量更大的队列中进行广泛性验证。同时可以考虑纳入更多可从外周血中获取的与肿瘤的发生、发展有关的信息进行研究,如可溶性血浆蛋白、CTC、细胞因子等。
1.1.4 多组学联用
以上研究中大多为单组学模型,无论是基因组还是转录组等都从分子层面深入挖掘了肿瘤免疫治疗中的基础生物学过程,具有重要临床意义。但任一单组学所能纳入的信息量有限,相较之下,多组学联合具有全面性、多维性、系统性等优势。Newell等[33]检测了77例接受抗PD-1治疗的晚期皮肤黑色素瘤患者肿瘤组织的全基因组、转录组、甲基化数据和免疫细胞浸润情况,通过回归算法逐步筛选变量,最终构建了基于γ干扰素(interferon-γ,IFNγ)和TMB的免疫治疗效果预测评分模型(AUC=0.84)。根据评分可将患者分为高应答者(评分>0.5)和低应答者(评分<0.5),这两组患者的PFS(P<0.000 1)及OS(P=0.001 8)差异均有统计学意义。该模型能以较高的精度预测免疫治疗效果(灵敏度为89%),但特异度欠佳,仅为53%,这可能与不同个体的耐药机制存在异质性有关。
2
通过影像组学数据结合机器学习来构建预测模型
影像组学是一种具备非侵入性、可重复性和高性价比的从医学图像中提取分子信息的方法。近年来,有研究[47]探索CT或18F-FDG正电子发射计算机体层成像(positron emission tomography and computed tomography,PET/CT)影像组学特征的临床用途,并将这些影像组学特征与肿瘤免疫治疗的应答情况结合分析,发掘具有免疫治疗效果预测价值的信息。
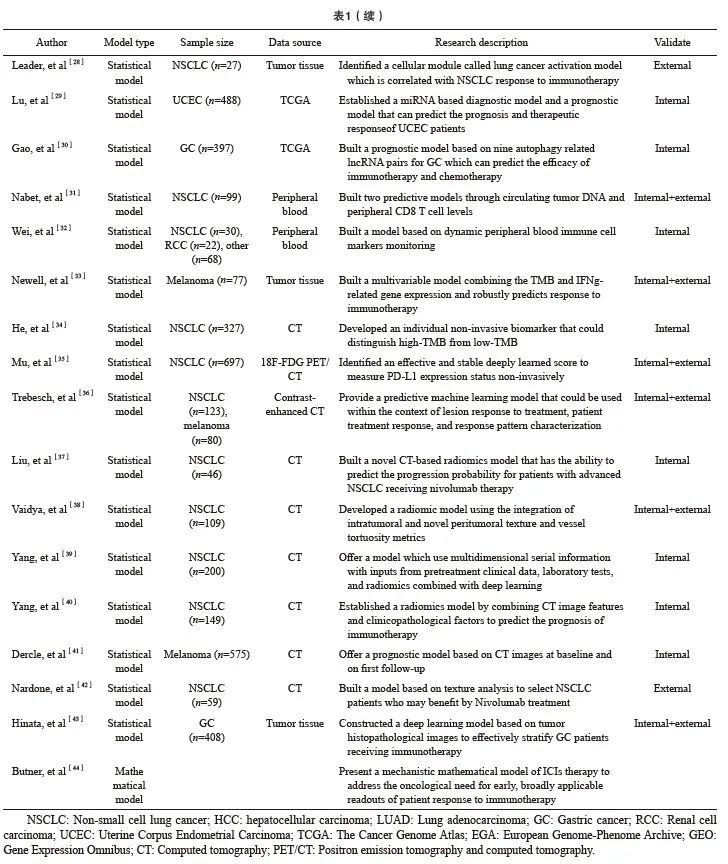
He等[34]共收集了327例NSCLC患者的CT影像数据和TMB数据,通过深度学习,挖掘出1 020个影像组学特征来区分高TMB和低TMB患者,并利用这些特征建立TMB放射性生物标志物(TMB radiomic biomarker,TMBRB),进而实现间接预测疗效。TMBRB可以通过无创方法获取,并有效地、低成本地将晚期NSCLC患者分组,低风险组较高风险组有更长的OS (301 d vs 533 d,HR=0.54,P=0.030)。与此类似,Mu等[35]收集了来自3个机构的697例NSCLC患者的18F-FDG PET/CT图像和临床数据,建立了PD-L1深度学习评分(deep learning score,DLS)来间接预测疗效,结果表明,PD-L1 DLS在PD-L1阳性和阴性患者之间的差异有统计学意义(AUC≥0.82,P<0.05),且在预测PFS和OS方面,DLS与免疫组织化学(immunohistochemistry,IHC)测量的PD-L1水平没有明显差异,表明PD-L1 DLS或可替代IHC来准确预测患者的免疫治疗临床获益。TMBRB和PD-L1 DLS虽然是间接预测指标,但两者都展现出良好的预测能力及临床可行性。而肿瘤影像组学特征是否与免疫治疗效果直接相关仍有待进一步探索。
通过获取NSCLC患者免疫治疗前的肺部CT影像,利用机器学习对病灶的大量影像组学特征进行提取计算,可以构建预测模型,将患者进行风险分类[36-39,42]。其中,Trebeschi等[36]发现具有不均匀密度模式和紧密边界的异质形态轮廓的病灶可能对免疫治疗的反应更佳,Nardone等[42]研究发现,病灶紧密度较低、纹理熵值高的患者往往预后更差,这些影像组学特征可能与肿瘤中央缺氧、坏死及高侵袭性有关。Yang等[40]在CT影像特征的基础上结合了临床病理学变量,建立了列线图模型,更利于临床决策。
然而针对此类模型,Dercle等[41]提出,只利用基线的影像组学数据并不能准确地预测患者预后,肿瘤大小的变化是反映癌症治疗效果的早期评估基础。因此,对比两次CT中患者病灶的变化能够为模型的建立提供新思路。该作者在研究中收集了575例晚期黑色素瘤患者的临床数据,并获取患者治疗前及治疗早期中的固定时间点的CT影像,利用机器学习随机森林算法评估免疫治疗患者的OS,最终筛选出4个影像组学变量:肿瘤的体积生长变化、肿瘤体积、肿瘤空间异质性的定量表征变化、肿瘤边缘表型的定量表征变化。由此确定了一组放射学特征,可以在治疗早期评估不可切除的黑色素瘤患者的OS。该模型经验证表现良好(AUC=0.92),证明了基线和首次随访时从常规CT图像中识别出的放射学特征可用于临床,为接受免疫单药治疗的黑色素瘤患者提供准确的预后预测。尽管本研究仅聚焦于晚期黑色素瘤患者,文中指出的4个预测特征中有3个已于另一项针对结直肠癌患者的研究[53]中得到验证,后续或可探索出具有普遍性的免疫治疗预后相关影像组学特征。
人工智能技术除应用于肿瘤放射学外,也可用于分析肿瘤组织的病理学图像。Hinata等[43]构建了基于胃癌组织病理学图像的深度学习预测模型(AUC=0.947),用以筛选出对免疫治疗敏感的人群。对于难以从放射学影像中获取较多有效信息的癌症类型,尤其是一些空腔脏器肿瘤,该方法更为经济且耗时较短,有助于对免疫治疗患者进行有效分层。
3
基于数理机制构建数学模型
通过开展对肿瘤免疫微环境及免疫药物与其相互作用机制的研究,现已积累了大量相关实验数据。使用系统和定量的方法,结合分子动力学、药代动力学、信号动力学、微环境适应机制等原理进行数学建模变得越来越重要,利用数理机制模型模拟肿瘤内生态环境及免疫药物在体内的生物学过程,可以帮助人们深入了解肿瘤的耐药机制,预测药物疗效[54]。
Butner等[44]通过数学方法还原了ICI干预下肿瘤负荷随时间变化的反馈过程,明确了这些生物过程之间的数学关系,建立了数学模型。作者从两次CT影像中获得肿瘤的生长速率,基于该数据,利用模型来量化肿瘤细胞杀伤率、抗肿瘤免疫状态、患者治疗后的肿瘤增长率,从而计算出肿瘤的总体负荷,最终据此来预测接受免疫治疗的转移性肝细胞癌及肺癌患者的结果。该模型能够较为准确地描述患者对ICI的不同反应模式,并识别出假性进展者。虽然利用肿瘤体积变化来预测疗效已有先例,但该研究首次从数学关系上描述了免疫治疗的多种生物学机制,并将其应用于前瞻性免疫治疗临床试验。
虽然利用数学方法建立疗效预测数理机制模型已经有了一定尝试,但此类模型缺乏临床验证,且鉴于人们对肿瘤免疫循环等生物学过程的机制研究仍然不够透彻,很多参数仍然未知,目前难以还原复杂的免疫系统全貌。
4
思考与展望
4.1 关于模型的评估与优化
为确保预测模型能够有效、精准,模型的建立应严格遵循数据收集与处理、变量筛选及模型构建、验证评估的流程。在此流程中,Poldrack等[55]提出用以建模的样本不应少于几百个,应严格对数据进行交叉验证,样本内模型拟合指数往往会夸大模型的预测能力,不应仅以此作为判断模型精准度的证据等观点。而本文所探讨的模型中,大部分的建模样本量都难以达到一定量级,而小样本数据往往去噪程度不足,易导致模型过拟合,泛化性不够,这是这类预测模型的普遍局限之处。常见的解决方法包括扩大研究规模、增大样本量和引入正则化算法等。
其次,利用回归算法建模,并最终形成风险评分系统是疗效预测模型常见的展现形式。此形式简单易理解,但需要对连续变量进行分类处理,会损失一定的预测准确度[56]。研究者应尝试并开发多元化的模型展现形式,包括列线图、图形评分表等。不同形式之间相互补充,能够更直观、有效地将预测模型与患者的临床决策相结合,提升预测准确度。
关于模型的验证,受数据量大小及方法的限制,部分研究缺乏内部交叉验证或未在外部独立数据集上进行验证。且不同的模型均在不同的人群中使用不同的测试集进行验证,缺乏模型之间的横向比较。为改善这一结果,需要在同一数据集上对多个模型进行测试,同时对比灵敏度、特异度和模型准确度等[16]。未来需要多中心、大规模的前瞻性研究来评估免疫治疗效果相关预测模型的有效性及实用性。
除改善模型的建立及评估方法外,我们也应着重关注相关领域最新的研究进展,对现有模型的参数进行动态调整,利用新的标志物来增益模型。同时,在单一组学模型的研究基础上,应尝试进行多组学联用,扩大数据的广度和维度,更系统地评估肿瘤微环境。
对于影像组学预测模型,目前此类研究的流程并不规范,不同中心收集的影像组学数据质量参差不齐,部分影像组学质量评分(radiomics quality score,RQS)较低,且此类研究难以建立影像组学生物标志物和生物学证据之间的因果关系,其内在机制仍需进一步探索。
4.2 临床应用
在实际的临床工作中,肿瘤基因检测费用较高,分析技术有一定门槛,模型的预测效能难以平衡其成本支出,因此很难取代目前临床常用的PD-L1检测。利用常规实验室检验数据,如血生化、血常规,以及CT、磁共振成像(magnetic resonance imaging,MRI)等低成本检测手段来开发模型有更高的临床可行性。对于基于影像组学数据的模型,虽然影像组学数据获取成本低,但后续利用人工智能辅助预测的过程中均需要专业医师手动分割病灶,分割过程会消耗大量的人力和时间,限制了模型的临床应用。目前,用于病灶自动检测和分割的深度学习技术正在开发中,这或许能够进一步优化该类模型,实现临床应用。值得关注的是,尽管利用人工智能进行疗效预测的准确度已十分可观,但是人工智能算法的不可解释性导致人们无法明晰其工作原理,这一直是限制人工智能在医疗领域实际应用的一大因素。因此如何规范计算机辅助的决策模式值得临床医学、伦理学等多学科共同探讨。
基于肿瘤组织标本的测序,虽然精确度、标准化程度高,可获取的信息丰富,但其样本获取难度及操作风险较大,难以在各级医院中普遍开展。近年来,更为微创的液态活检备受关注。液态活检是基于体液样本的非侵入性的检测方法,在肿瘤免疫治疗领域中以外周血为基础的液态活检样本最为常见。通过患者的血液样本获取游离的CTC、ctDNA、外泌体等,可以对肿瘤的演变进行动态评估[57-58]。但液态活检也面临检测流程复杂、假阴性率高等问题,未来深入挖掘基于外周血样本的数据,并结合相关指标建立预测模型值得继续探索。
在大数据时代,建立临床诊疗的全程化预测系统已经成为趋势,对于肿瘤免疫治疗我们同样可以进行尝试。随着免疫治疗的广泛应用,肿瘤患者的生存率得到了提升,但有70%~91%的患者会发生免疫相关不良事件(immune-related adverse event,irAE),可累及全身多种器官和组织,包括皮肤、甲状腺、肝、肺等[59]。此外,免疫治疗过程中出现耐药、超进展、假性进展等情况均会影响肿瘤患者的临床获益。因此,今后的研究可以考虑在疗效预测模型中增加包括irAE在内的多种结局事件作为探索指标,以形成抗肿瘤免疫治疗的全程化精准预测整合平台。
利益冲突声明:所有作者均声明不存在利益冲突。
[参考文献]

本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言