Clin Chem:基于脑脊液cfDNA的纳米孔测序可对脑肿瘤患者进行初步诊断及分类
2023-09-17 测序中国 测序中国 发表于上海
研究团队对来自脑肿瘤患者脑脊液(CSF)中的游离DNA(cfDNA)进行了纳米孔测序,并使用随机森林分类器分析了拷贝数变异(CNV)和整体DNA甲基化模式。
中枢神经系统(CNS)肿瘤是原发或继发于脑和脊髓的一组良恶性疾病,具有很强的异质性,可分为100多种不同的类型。明确诊断肿瘤的确切类型和亚型,对于制定合适的治疗计划至关重要,但目前脑肿瘤的分子诊断通常依赖于组织活检,这可能会带来麻醉或神经外科手术相关的诸多风险。此外,在初步诊断后,如需进一步评估肿瘤进展、复发或获得新的基因改变,只能通过重新活检来证明。
近年来,测序技术和DNA甲基化分析发展迅速,已成为评估不同肿瘤类型的有利工具。通过测序技术可识别肿瘤的诊断标志物或可靶向的改变,但由于部分肿瘤可能不具有特定的单核苷酸变异或基因融合的特征,因此仅依据测序很难对其进行可靠的预测。基于完整的DNA甲基化分析,能够评估基因组内的数千个CpG位点,从而获得肿瘤的多维特征;将这些特征与参考数据库进行比较,可确定样本与特定CNS肿瘤实体的最高相似性。
近日,德国汉堡-埃彭多夫大学医学中心的研究人员及合作者在Clinical Chemistry上发表了题为“Classification of Brain Tumors by Nanopore Sequencing of Cell-Free DNA from Cerebrospinal Fluid”的文章。研究团队对来自脑肿瘤患者脑脊液(CSF)中的游离DNA(cfDNA)进行了纳米孔测序,并使用随机森林分类器分析了拷贝数变异(CNV)和整体DNA甲基化模式。结果显示,在45%的样本中检测到了循环肿瘤DNA(ctDNA)并成功对肿瘤进行了分类,包括疾病进展患者和无已知残留病变患者样本。在所有含有可检测肿瘤DNA的样本中,CNV分析成功率高于比甲基化分析,总检出率分别为88%和44%。综上,来自CSF样本的cfDNA纳米孔测序数据可能是脑肿瘤初步诊断的一种潜在方法,也是疾病监测的重要工具。

文章发表在Clinical Chemistry
对CNS肿瘤进行精确的分子诊断,通常需要通过手术获得肿瘤组织;事先了解肿瘤类型可能会改进手术策略并减少手术相关风险。目前,液体活检分析越来越受到关注,已广泛用于各种癌症,其分析物(例如循环CTC和cfDNA)已被纳入临床试验研究。对于CNS肿瘤而言,CSF仍是诊断和监测的首选;cfDNA是一种相对稳定的CSF分析物,可通过全基因组低覆盖测序重现肿瘤的拷贝数变化,并可作为微小残留病的预后标志物。
研究团队首先采用低覆盖度纳米孔测序,对来自脑肿瘤患者129个CSF样本中的cfDNA进行了分析,并使用随机森林分类器(NanoDx)分析了CNV和甲基化模式,这些样本来自99名患者,共涵盖22种肿瘤。其中,髓母细胞瘤数量较多(38%),其次是室管膜瘤和毛细胞星形细胞瘤。患者中位年龄为19.2岁,大多数患者是儿童或青少年。
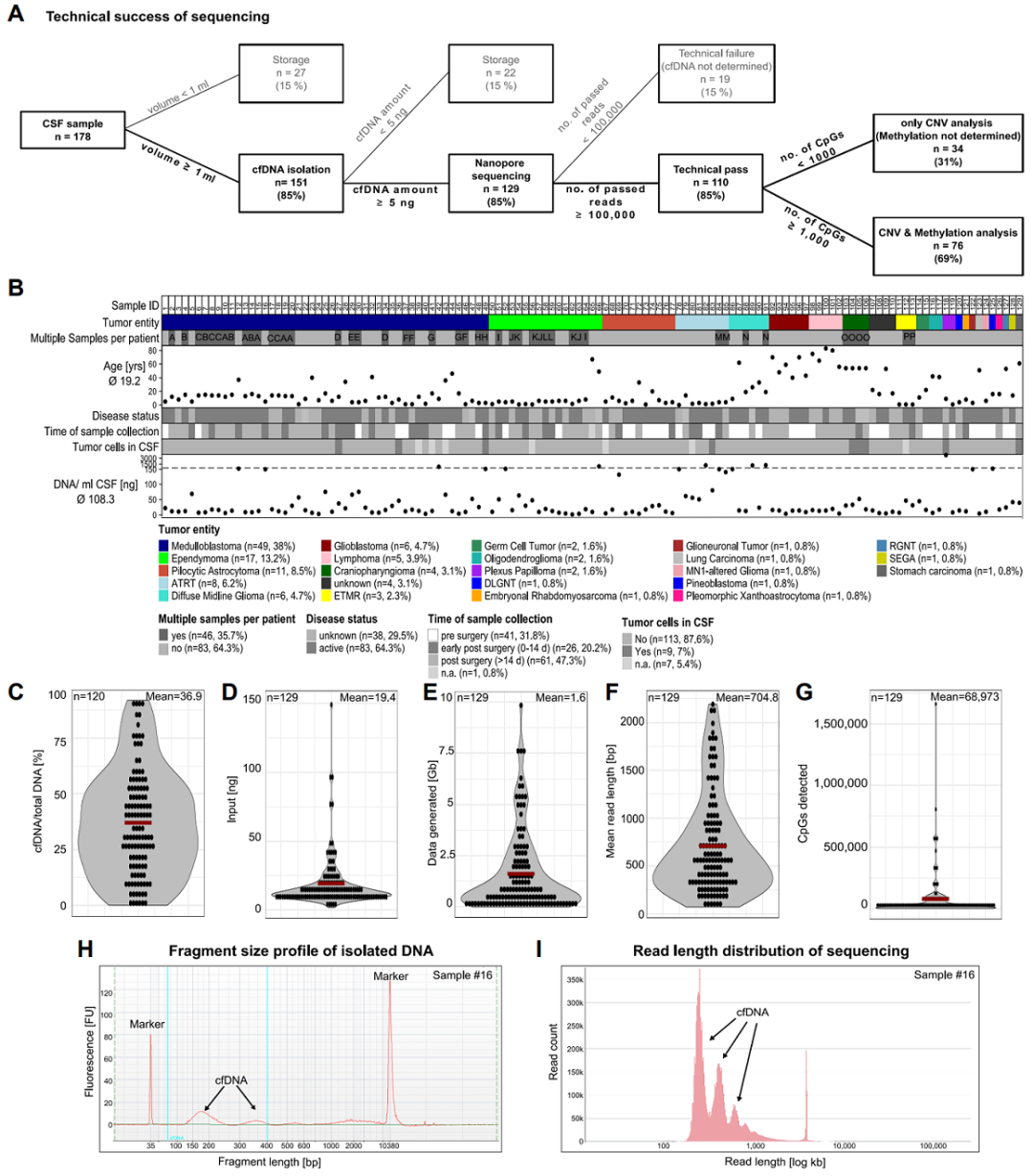
图1. 样本队列和技术特征概述。
结果显示,有110个样本被成功分析,其中有50个样本(45%)通过CNV或甲基化分析检测到ctDNA,这些样本包括术前和术后早期样本以及术后>14天样本,通常伴有临床未知的残余肿瘤或疾病复发。在22%的样本中,仅由CNV分析检测到ctDNA;在18%的样本中,同时由CNV和甲基化分析检测到ctDNA;在5%的样本中,仅由甲基化分析检测到ctDNA。
当NanoDx评分为0.072、均匀流形近似和投影(UMAP)可视化中胶质母细胞瘤聚类清晰时,两种分析方法均对肿瘤样本进行了正确分类。此外,研究团队还通过CNV分析发现了一些肿瘤组织或CSF特有的诊断、预后改变,例如多层菊形团样胚胎性肿瘤(ETMR)中C19MC的扩增、后颅窝A组(PFA)室管膜瘤中chr1q的增益及chr6q的减少,这些都是重要的预后标志物。
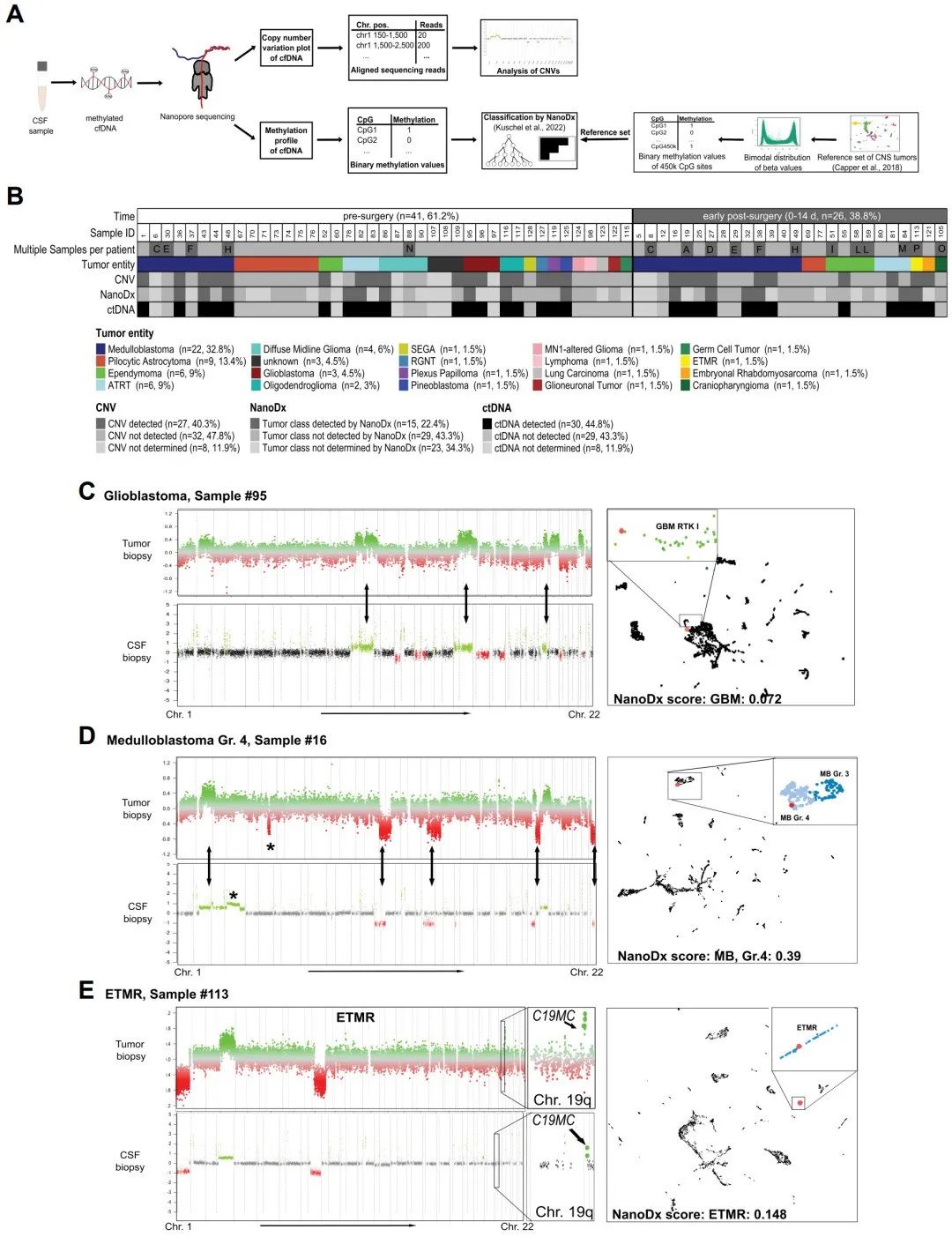
图2. 术前和术后早期样本的拷贝数和甲基化分析。
接下来,研究团队对手术后采集的61个样本进行了分析(图3)。结果显示,50个样本(82%)完成纳米孔测序;在20个样本(32.8%)中,ctDNA可以通过至少一种方法被检测到。CNV分析显示,17个样本(27.9%)中存在ctDNA。甲基化分析仅在7个样本(11.5%)中实现了正确的肿瘤分类。
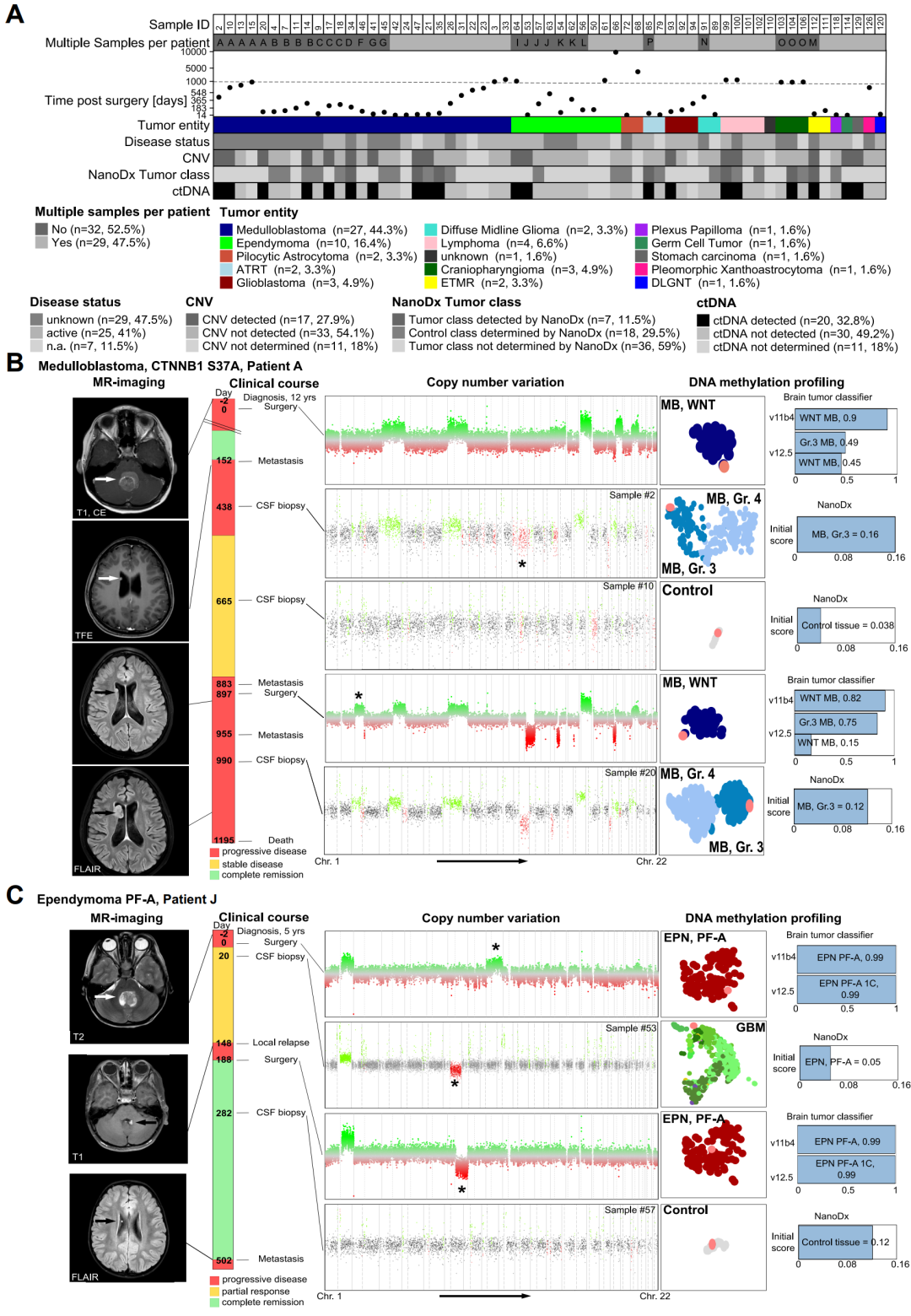
图3. 术后和整个病程样本的拷贝数和甲基化分析。
上述结果表明,在50/129个样本(39%)和50/110个技术上成功的样本(45%)中,可以通过至少一种分析方法检测到ctDNA(图4)。研究团队还将上述分析结果与肿瘤组织的临床诊断和分子分析(如有)进行了比较,发现大多数CNV特征与相应肿瘤组织的特征一致。
此外,在检测到ctDNA的样本中,只有5个样本含有可在显微镜下检测到的肿瘤细胞。最后,研究团队进行了纵向分析,发现了与肿瘤复发有关的CNV,这凸显了液体活检检测肿瘤生物学潜在相关变化的潜力。
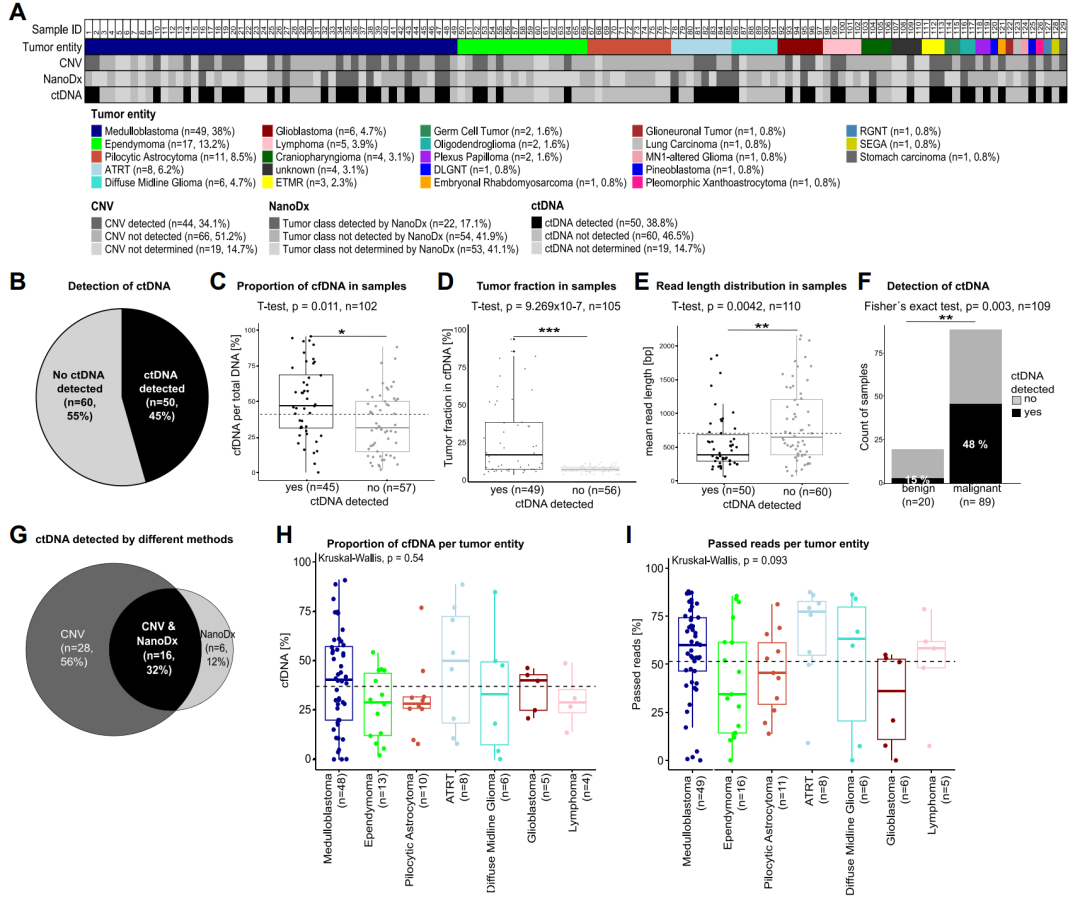
图4. cfDNA测序CNV和甲基化分析总结。
综上所述,该概念验证研究表明,纳米孔测序是通过CSF液体活检诊断CNS肿瘤的有力工具,能够在不同肿瘤实体、疾病状态的良性及恶性脑肿瘤样本中检测到ctDNA。与潜在肿瘤细胞的显微镜检查相比,基于cfDNA的液体活检使严密和纵向的肿瘤监测成为可能,具有额外的临床价值。总之,用于诊断和监测脑肿瘤的脑脊液cfDNA纳米孔测序有望整合到常规临床流程中,帮助医生实现高效、准确的诊断,并减轻患者医疗负担。
参考文献:
Afflerbach AK, Rohrandt C, Brändl B, et al. Classification of Brain Tumors by Nanopore Sequencing of Cell-Free DNA from Cerebrospinal Fluid [published online ahead of print, 2023 Aug 25]. Clin Chem. 2023;hvad115. doi:10.1093/clinchem/hvad115
https://academic.oup.com/clinchem/advance-article-abstract/doi/10.1093/clinchem/hvad115/7251226?redirectedFrom=fulltext&login=false#no-access-messag
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言











