PNAS:复旦大学王应祥团队揭示减数分裂异染色质浓缩的分子机制
2022-10-24 复旦大学 复旦大学
该研究揭示了多面手DNA聚合酶POLε参与减数分裂异染色质形成的分子机制,证明其参与DNA合成的同时,可能通过催化亚基POL2A的C端识别并参与组装组蛋白H3.1-H4形成核小体。
减数分裂是真核生物有性生殖所必需的生命过程,其经过同源染色体重组,非同源染色体自由组合和两次分裂最终形成单倍体配子,即保证了遗传物质的相对稳定,又增加了个体间的差异,使物种得以繁衍和进化。减数分裂与农作物育性和产量,以及人类生殖健康息息相关。
染色体由相对松散的常染色质和高度浓缩的异染色质组成。相对于常染色质基因相对富集,而异染色质区域重复序列和转座子相对富集。减数分裂中,常染色质区域是重组的发生热区,而异染色质则是重组发生的冷区。此外,正常异染色质的形成对抑制转座子的活性,维持基因组的稳定性和保证染色体正确分离至关重要。目前,在有丝分裂过程中异染色质形成和浓缩的机制已有一定认知,而在减数分裂中仅了解到异染色质区域抑制重组的发生和对早期同源染色体配对有促进作用,其染色质的动态变化和功能性质都有别于有丝分裂过程,但减数分裂中异染色质的形成和浓缩机制尚不清楚。
2022年10月17日,复旦大学王应祥团队在PNAS在线发表了题为“DNA Polymerase Epsilon Binds Histone H3.1-H4 and Recruits MORC1 to Mediate Meiotic Heterochromatin Condensation” 的研究论文。该研究揭示了植物减数分裂过程中 DNA聚合酶epsilon(POL ε)特异识别组蛋白二聚体H3.1-H4并招募MORC1蛋白控制异染色质形成的分子机制。
课题组前期研究发现DNA前导链合成酶POL ε的最大亚基POL2A在减数分裂重组中具有重要功能(Huang et al., PNAS, 2015),但是其潜在的分子机制还不清楚。最近的研究已经揭示了POL2A在常染色质的重组热区,通过招募SUVH2/9抑制基因转录的分子机制(Wang et al., PNAS, 2022)。
有趣的是,该基因突变也影响了减数分裂中异染色质的正常浓缩。在该研究中,研究者运用细胞学分子标记证明pol2a突变体中异染色质结构和修饰异常;为了探究作为DNA复制因子的POL2A如何调控异染色质结构,研究者通过互作筛选发现POL2A的N端可以和浓缩蛋白ATPase MORC1相互作用,并共同影响减数分裂异染色质结构和修饰,暗示了它们可能处于同一遗传通路中;同时研究者还发现减数分裂时期MORC1在染色体上的定位依赖于POL2A,暗示POL2A在减数分裂时期负责招募MORC1到异染色质参与浓缩过程。
而作为 DNA聚合酶,POL ε又是如何招募MORC1到异染色质区?研究者进一步研究发现POL2A的C端锌指(ZF)结构域可以特异地结合组蛋白H3.1-H4的四聚体,并且在不同物种中保守。之前的研究发现组蛋白变体H3.1通常在异染色质富集,对于异染色质形成和修饰都至关重要,并且其特异的在DNA复制时期通过CAF1复合体装载到染色体上。研究者通过转基因遗传分析显示POL2A的ZF结构域对于异染色质结构和H3.1的定位都至关重要;并且有意思的是组蛋白变体H3.1上的两个特殊氨基酸A31和S87突变会影响和POL2A-ZF的互作,并且也会影响自身在植物体内定位。这些结果暗示了POL2A通过特异识别H3.1-H4,可能直接参与到DNA复制偶联的H3.1核小体组装中并定位到异染色质区。
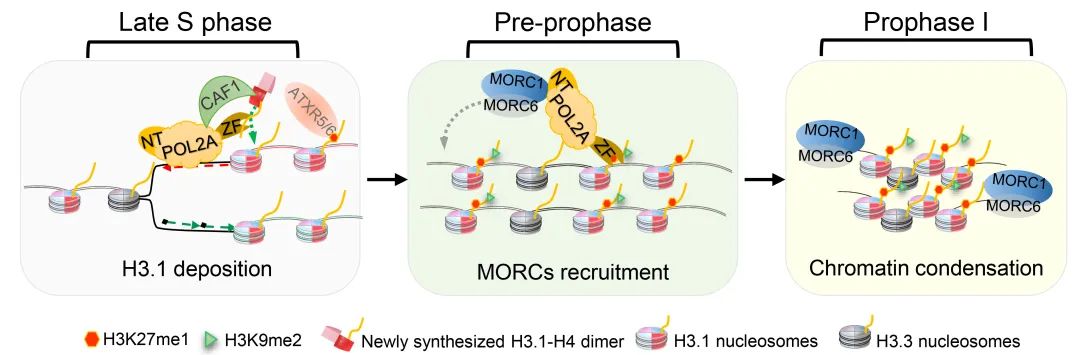
图1. POL ε调控减数分裂异染色质形成模型
综上所述,该研究揭示了多面手DNA聚合酶POLε参与减数分裂异染色质形成的分子机制,证明其参与DNA合成的同时,可能通过催化亚基POL2A的C端识别并参与组装组蛋白H3.1-H4形成核小体;复制完成后POL2A通过结合H3.1-H4继续停留在异染色质区并通过N端招募浓缩复合体MORC1/6,最终促进减数分裂高度浓缩的异染色质形成(图1)。由于POL ε在不同生物中高度保守,研究者推测该研究发现的其在减数分裂中的作用机制在真核生物中相对保守。
最后,值得一提的是王应祥团队在PNAS发表的两篇文章比较全面地揭示了DNA聚合酶POLε在染色体复制后进入减数分裂,其分别结合在减数分裂常染色质和异染色质区域的新功能。
复旦大学生命科学学院博士后王聪为两篇文章的第一作者,北卡罗来纳大学教堂山分校博士后/华南农业大学教授黄霁月为本篇论文的并列第一作者,复旦大学/华南农业大学/岭南现代农业生物与技术广东省实验室王应祥教授,美国北卡罗来纳大学教堂山分校Gregory Copenhaver和美国宾夕法尼亚大学生物系马红教授为共同通讯作者。复旦大学董爱武教授和中国科学院大学/中国科学院遗传与发育生物学研究所姜丹华研究员提供了指导和帮助,复旦大学研究生李颖平、张俊、何承鹏和李天杨也参与了该项工作。相关工作得到了国家自然科学基金委、复旦大学、中国博士后基金和北卡罗莱纳大学的资助。
文章链接:
https://www.pnas.org/doi/10.1073/pnas.2213540119
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言