Scientific reports:CRISPR检测新方法
2023-07-10 小桔灯网 小桔灯网 发表于上海
本文介绍的工作建立在高通量Cas13a crRNA筛选的工作基础上,旨在发现crRNA分子的共同特征。
传染病病原的快速和特异性检测的金标准是基于核酸的检测方法,如聚合酶链反应(PCR),但成本高、仪器昂贵且需要训练有素的人员。基于CRISPR-CAS系统的检测方法最近作为基于PCR诊断的替代方法出现。CRISPR检测依赖于crRNA识别特定DNA (DETECTR)或RNA (SHERLOCK)靶标的最佳“设计规则”。
最近,Metsky等报道了一种基于神经网络的机器学习(ML)算法,根据大规模Cas13a crRNA旁切核酸酶筛选获得的数据进行训练,自动设计针对大量不同病原体的靶向crRNA。该方法主要依赖于选择多个特定的引导RNA,通常耐受≤2个间隔区错配,以覆盖广泛的靶标范围。
巴恩斯等人最近报道了用于病毒检测的简并crRNA(degenerate crRNAs)的概念。在Li等另一项研究中,研究人员开发了一种使用的简并crRNA来检测克里米亚-刚果出血热病毒(CCHFV)多谱系的方法。该研究表明,使用的简并crRNA与密切相关的病毒没有交叉反应,可以耐受多达3次错配,并且仍然激活Cas13a酶的旁切活性。
一些研究小组已经应用ML模型来协助基于cas9的CRISPR应用的指导设计。近日,一组来自于美国的研究团队在杂志Scientific reports上发表了一篇题为“Machine learning for design of degenerate Cas13a crRNAs using lassa virus as a model of highly variable RNA target”的文章。本文介绍的工作建立在高通量Cas13a crRNA筛选的工作基础上,旨在发现crRNA分子的共同特征。最初的目标是通过实验确定最小数量的简并crRNAs,可以激活Cas13a旁切活性。随后,作者应用RuleFit决策树机器学习算法来寻找决定Cas13a核酸酶活性的间隔序列属性。

图片来源:Scientific reports
主要内容
简并crRNA间隔序列的性能
第一步是通过经验观察哪些crRNAs可能允许基于cas13的高效检测位于L和GPC基因中的LASV靶序列。简并序列是在简并位置上具有备选核苷酸的序列的所有排列的混合物。采用两种简并核苷酸:R (A/G)和Y (C/T)。对四组简并crRNA集进行测试,为LASV谱系II和谱系IV序列设计单独的crRNA集(下图A)。
通过测量在3nM靶RNA存在下2小时内Cas13a测定中产生的总荧光信号来评估crRNA的性能。测试结果显示,大多数crRNA(根据目标序列的不同,从82%到93%)被归类为表现出高或中等性能,只有一小部分crRNA(从7%到18%)不产生信号或低信号。这些结果表明,简并crRNA具有与常规crRNA相似的性能。
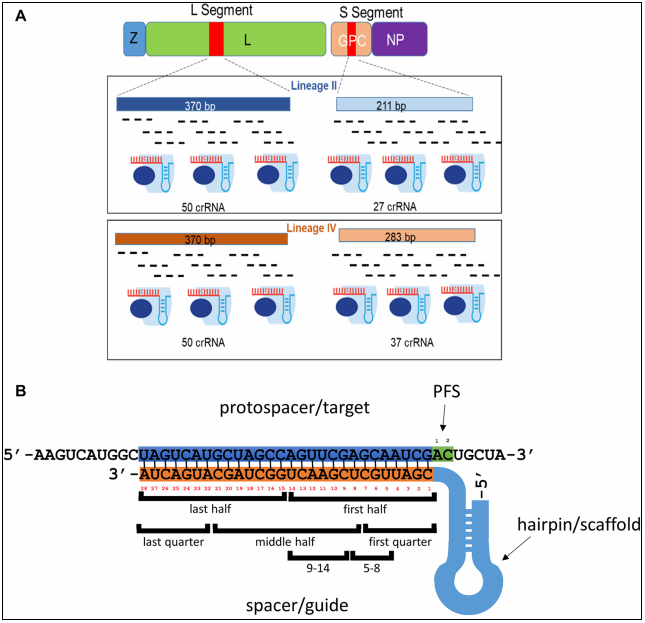
简并crRNA间隔序列的设计。图片来源:Scientific reports
高性能crRNA的检测极限和脱靶活性
每种简并crRNA都含有多个具有可变间隔序列的crRNA分子。作者进行了多次实验,以了解这种高水平复用是否会导致非特异性序列的检测,以及它如何影响检测的检测限(LOD)。结果显示,非特异性靶标均未产生任何高于无靶标阴性对照的信号,表明缺乏脱靶活性。
为了确定LOD,对crRNA进行Cas13a活性测定,使用10倍靶稀释系列,范围为3 nM至3 pM。最低可检测目标浓度被确定30-300 pM。这些结果与先前获得的非简并crRNA的LOD相当。且在基因组RNA背景存在的情况下,信号减少相对较小(目标浓度为30pm时荧光信号减少约2倍),表明背景RNA并不会产生很大干扰。
LASV谱系和LASV近邻检测
为了确定简并crRNAs可以检测的LASV谱系范围及其与邻近非LASV序列的潜在交叉反应性,作者选择了8种设计用于检测L靶标的高性能crRNAs,并使用Cas13a活性测定对这些crRNA进行测试。对这些crRNA进行了12个代表已知的所有LASV谱系(I至VII),和11个与LASV密切相关的近邻旧大陆沙粒病毒(OWA)的靶序列的测试。结果总结在下表中。crRNA #5_LIV被发现是区分LASV及其近邻的最佳crRNA,11个近邻中只有一个被分类为阳性。总的来说,crRNAs #5_LII、#5_LIV、#9_LII和#9_LIV对LASV和邻近靶标具有较好的区分能力。这些结果表明,检测到的靶标范围与特定crRNA的特定序列有关,并且在某些情况下允许检测所有测试的LASV谱系。
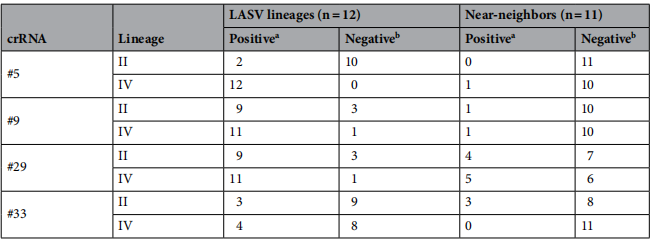
LASV谱系及其近缘系L基因的检测
图片来源:Scientific reports
间隔序列/靶标配对属性对Cas13a活性的影响
所有8个被测试的简并crRNA间隔序列与所有靶序列比对,以确定错配的数量和位置对Cas13a活性测定结果的影响。下图绘制了所有测试的crrna与荧光信号不匹配的数量。可以看出,错配总数与荧光信号的平均强度呈负相关关系。大多数具有三个或更少错配的间隔/目标对产生了足够的荧光信号,将其分类为阳性。对于有5个或更多错配的间隔/目标对,荧光信号被分类为阴性(只有一个例外),而恰好有4个错配的间隔物/目标对包含被分类为阳性和阴性的组合。
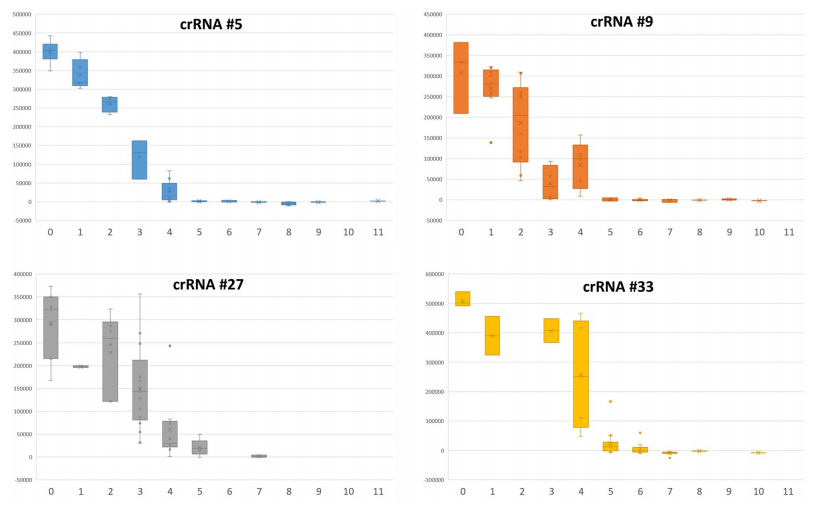
间隔序列/靶标配对特性对Cas13a活性的影响
图片来源:Scientific reports
机器学习预测信号强度
为了了解除错配总数以外变量对信号强度的影响,作者开发了基于RuleFit算法的预测模型。RuleFit分类器经过训练,使用三个错配数据集来预测引导RNA/目标对是否会产生高于或低于阈值的信号。Watson-Crick配对数据集经过十次交叉验证后的输出精度为95.0%±2.0%,AUC为0.97。
使用该模型,作者研究了特征重要性的规则。对所有三个数据集的预测总体影响最大的特征是总错配计数(n)。第二个最重要的特征是四分位距(IQR),它反映了跨间隔的错配分布。对于Watson-Crick配对,5′端错配的位置(min)和位于原间隔侧翼1号位点(PFS_1)的核苷酸的也会影响预测(如下图)。总体而言,分析特征的性质表明,虽然总体错配计数(n)是最重要的,但这些错配的位置和分布以及PFS #1上核苷酸的属性对分类(即检测结果)的相对影响最大。
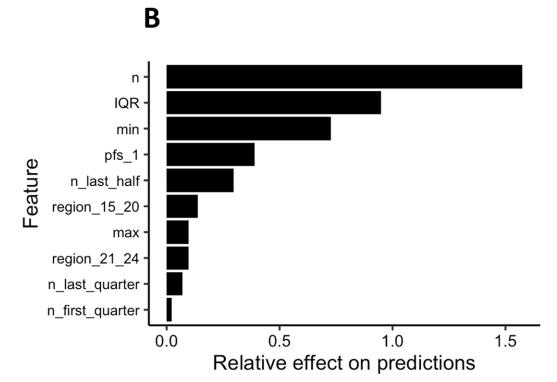
特征对模型预测的相对影响。图片来源:Scientific reports
预测规则
用于生成预测规则的决策树示例如下图所示。使用的特征是n、IQR、min和PFS_1。使引导RNA信号最大化的决策规则是:
1) 如果IQR≥6.25, n在0到3之间,pfs_1为A、U或C;
2) 如果IQR < 6.25, n在0到2之间;
3) 如果n = 4, IQR在9到28之间,min在1到3之间。
这些规则可从44%的初始池(包括最多4个错配的引导RNA)中获得95%的阳性引导RNA。
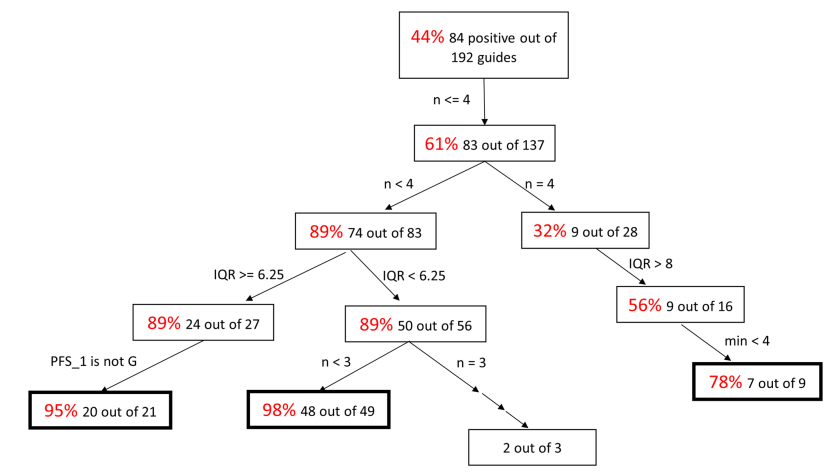
决策规则总结。图片来源:Scientific reports
讨论和结论
本研究的目的是利用基于Cas13a的CRISPR检测方法,评估具有简并间隔序列的crRNA在检测高度可变RNA靶标方面的效用。作者测试了设计用于检测不同RNA靶标的候选简并Cas13a crRNA集。应用决策树机器学习(ML)算法(RuleFit)来定义决定简并crRNA引发旁切活性特异性的特征。虽然错配的总数(0-4)很重要,但其特异性也取决于错配的间距,以及它们与间隔5 '端的接近程度。作者开发了一种用于设计候选简并crRNA集的预测算法,允许更好地区分“包括”和“排除”相关靶序列组。此通用ML方法可以应用于任何CRISPR/Cas系统的简并crRNA集的设计。
本研究中提出的基于RuleFit的分类器仅使用由LASV及其近邻表示的一个高度可变目标的序列进行验证。为了确定该算法不偏向于LASV序列中存在的特定模式,并且更普遍地适用,将需要对其他高度可变的目标进行测试。该研究的另一个局限性是,只有具有相对固定组成(恒定间隔和发夹长度)的引导物才能筛选其激活LwaCas13a旁切活性的能力,其激活能力大于主观定义的阈值。特定应用可能需要更短的反应时间以及不同的主观截止阈值。原则上,使用本文描述的类似实验和ML方法可以很容易地实现ML对不同CRISPR/Cas蛋白组合的应用。
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言








传染病病原的快速和特异性检测的金标准是基于核酸的检测方法
48
基于CRISPR-CAS系统的检测方法最近作为基于PCR诊断的替代方法出现。CRISPR检测依赖于crRNA识别特定DNA (DETECTR)或RNA (SHERLOCK)靶标的最佳“设计规则”。
33