Nat Commun:FinaleMe模型:基于血浆cfDNA片段化模式预测DNA甲基化水平和起源组织状态
2024-04-29 测序中国 测序中国 发表于陕西省
研究团队开发了一种名为FinaleMe的计算方法,可预测每个cfDNA片段中每个CpG的DNA甲基化状态,并获得CpG位点的连续DNA甲基化水平;进一步从推断的甲基化模式中预测相关的起源组织状态。
DNA甲基化是最早被发现、也是研究最深入的表观遗传调控机制之一,在疾病进展和胚胎发育过程中起着重要作用。细胞游离DNA(cfDNA)中的全基因组DNA甲基化水平已被广泛研究,可用于疾病诊断及预后。目前,亚硫酸氢盐测序(BS)仍是检测cfDNA甲基化的金标准,但其处理过程中会造成DNA片段的非均匀序列依赖性降解,导致input DNA的大量丢失,从而限制了诊断测试和分析的灵敏度。
与基因组DNA(gDNA)不同,cfDNA不是随机片段化的,其片段化模式与局部表观遗传背景高度相关。已有研究发现,甲基化和非甲基化cfDNA分子之间的DNA片段模式存在显著差异,这提示了从cfDNA片段模式计算推断DNA甲基化水平的可能性。近期一项研究提供了一个概念验证解决方案,通过深度学习模型预测高覆盖率WGBS中DNA甲基化的二元状态,但cfDNA全基因组测序(WGS)中预测甲基化状态的能力仍有待探索。
为解决上述难题并充分利用cfDNA WGS数据集,美国麻省理工学院和哈佛大学Broad研究所的科研人员在Nature Communications发表了题为“FinaleMe: Predicting DNA methylation by the fragmentation patterns of plasma cell-free DNA”的研究文章。研究团队开发了一种名为FinaleMe的计算方法,可预测每个cfDNA片段中每个CpG的DNA甲基化状态,并获得CpG位点的连续DNA甲基化水平;进一步从推断的甲基化模式中预测相关的起源组织状态。最后,研究团队使用80对深度(~16-39×)和低深度(~0.1×)WGS和WGBS数据验证了FinaleMe性能。

文章发表在Nature Communications
由于DNA甲基化与核小体占有率密切相关,研究团队假设cfDNA片段的边界可能因其与核小体的结合而存在偏差,那么在每个cfDNA分子中观察到的片段模式揭示其相关的DNA甲基化模式,从而表明其起源组织。为验证这一假设,研究团队首先分析了两个健康个体cfDNA和buffy coat(白膜层细胞)样本gDNA中片段大小与DNA片段平均甲基化水平之间的相关性,其中cfDNA从公开可用的WGBS中获得(图1)。结果显示,cfDNA重复样本在单核小体长度上显示出波动的甲基化模式,这一模式在gDNA样品中不存在。上述结果支持了cfDNA的片段化模式可以提供与DNA甲基化水平相关的信息。
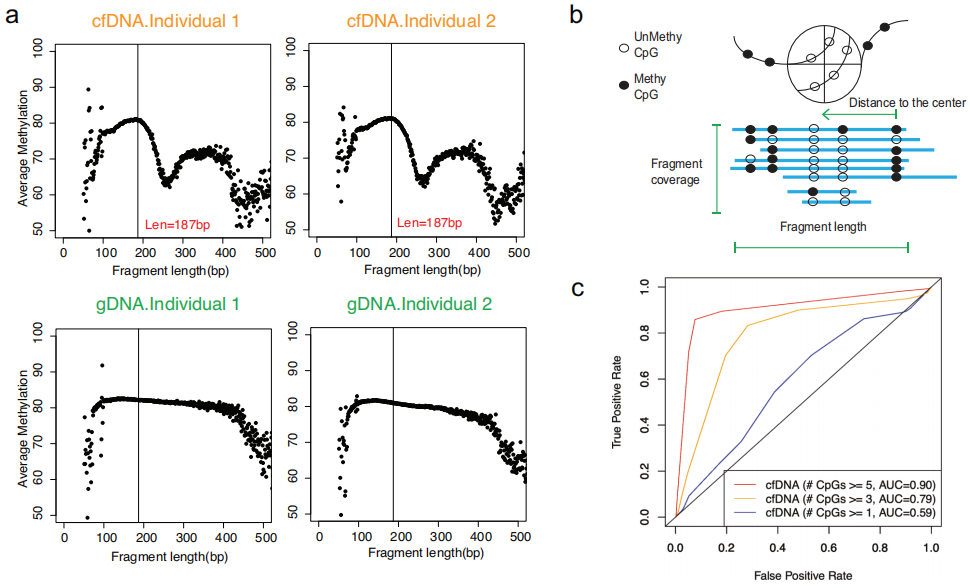
图1.从高覆盖率全基因组亚硫酸氢盐测序推断DNA甲基化
接下来,研究团队够建了一个名为FinaleMe的非均匀隐马尔可夫模型(HMM),用于预测cfDNA中的甲基化状态(图2)。鉴于CpG在人类基因组中的分布并不均匀,该模型考虑了CpG位点之间的距离,并利用片段长度、归一化覆盖率和每个CpG到DNA片段中心的距离作为特征(图1b)。研究团队首先使用高覆盖率WGBS(掩盖甲基化状态)对模型进行评估,然后使用每个DNA片段中每个CpG的真实DNA甲基化状态对模型性能进行基准测试。
在对等量的甲基化和非甲基化CpG进行采样后,研究团队观察到,根据CpG丰富区域内的受试者操作特征曲线下面积(auROC),可从每个DNA片段中预测每个CpG的甲基化状态,预测性能良好,当片段中CpG数量≥5个时,auROC为0.91。
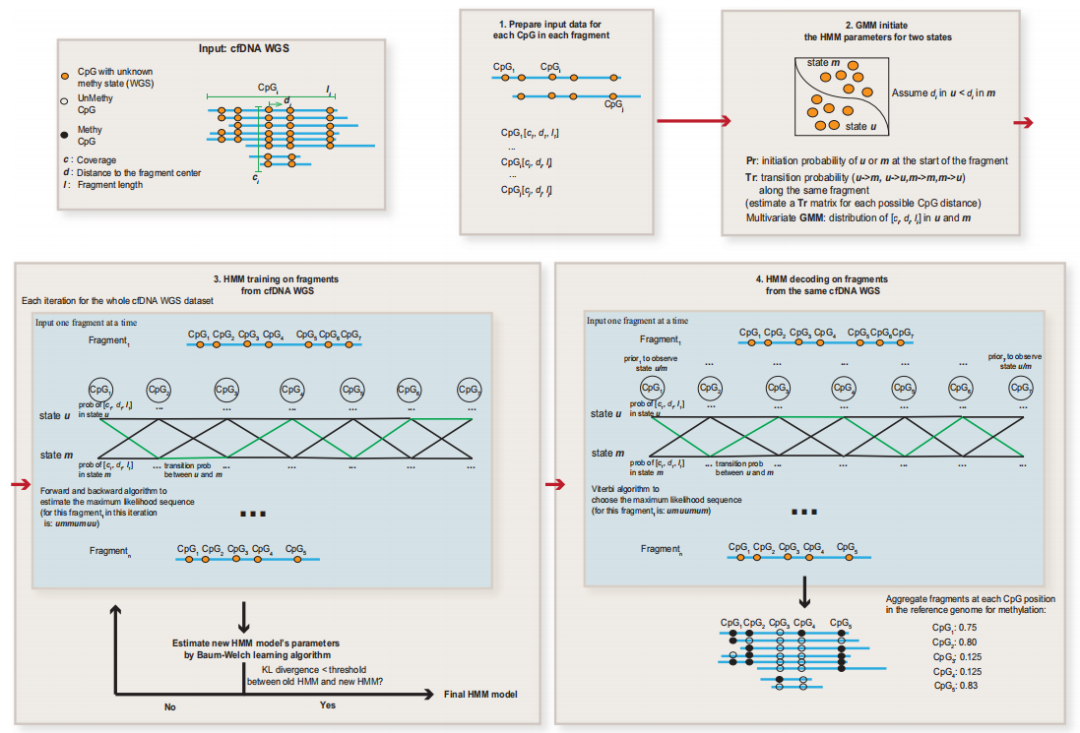
图2. FinaleMe模型总结
为进一步衡量FinaleMe模型在cfDNA WGS中的性能,研究团队从健康个体和前列腺癌患者同一管血液的血浆cfDNA样本的生成了高覆盖率WGS和WGBS数据(图3)。在不使用cfDNA WGBS数据的情况下,研究团队训练了FinaleMe模型并使用相同cfDNA WGS 数据集预测了甲基化水平。通过将这些结果与相匹配WGBS参考基因组中CpG位点的甲基化水平进行比较,发现在CpG富集区域的单CpG和1 kb窗口处存在高度相关性。此外,在癌症和健康个体CpG富集区域的差异甲基化区域(DMR)中,匹配cfDNA WGS预测的甲基化水平出现一致性变化。
为检验该模型的潜在过拟合问题,研究团队进一步训练和解码了来自癌症和正常血细胞的gDNA WGS模型。gDNA WGS的预测结果显示,在相匹配gDNA WGBS数据集检测到的DMRs中,癌症细胞和正常细胞的甲基化无差异,这表明在cfDNA WGS中预测的差异甲基化是由片段化特征驱动。此外,与CpG富集区域相比,FinaleMe在CpG缺乏区域的预测性能较差。
研究团队进一步评估了重要调控元件的甲基化水平,如CpG岛(CGI)启动子、5'外显子边界和CTCF基序。结果显示,在健康个体和癌症患者cfDNA中,真实数据(WGBS)和预测值(WGS)之间存在高度相关性,但在gDNA数据集中未发现这种相关性。
由于CGI和CGI shore区域的DNA甲基化通常是细胞类型特异性的,研究团队通过使用WGBS和WGS检测或预测的DNA甲基化水平进一步估计了cfDNA中的起源组织。在癌症和健康条件下,每个个体的预测和检测甲基化水平之间具有相似的组织起源特征(图3f),这与先前研究结果基本一致。
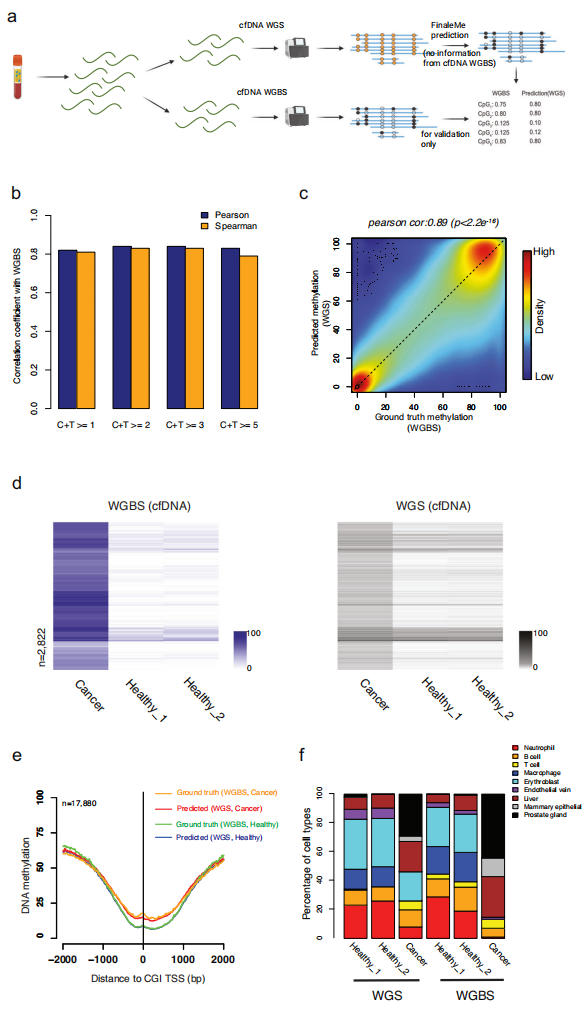
图3.从高覆盖率全基因组测序推断DNA甲基化
深度WGBS和WGS在常规临床应用中成本昂贵,为此,研究团队探究能否通过超低深度全基因组测序(~0.1×,ULP-WGS)预测DNA甲基化水平,从77名个体(包括健康个体、乳腺癌和前列腺癌患者)中生成了相匹配的ULP-WGS和超低深度WGBS(ULP-WGBS)cfDNA数据。
研究团队检测了全局和重要调控元件的甲基化水平,观察到ULP-WGS和ULP-WGBS预测的甲基化水平和实际检测的甲基化水平分别具有相似的平均甲基化特征;在DMR中,ULP-WGS和ULP-WGBS检测的甲基化水平不同。此外,利于ULP-WGS和ULP-WGBS的甲基化水平可用于预测起源组织,这两种检测的结果一致,其与基于深度测序预测的结果也基本一致。这些结果表明,在DNA甲基化和起源组织预测方面,将FinaleMe应用于ULP-WGS中可得到与ULP-WGBS一致的预测结果。
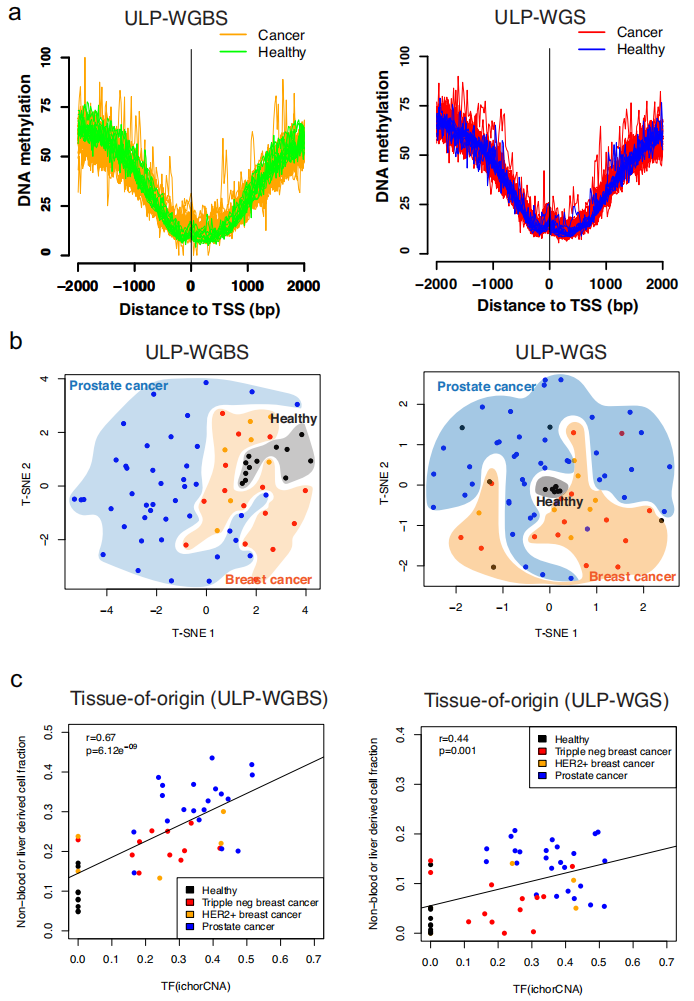
图4.从cfDNA ULP-WGS推断DNA甲基化和起源组织
综上所述,该研究开发了FinaleMe,可通过分析cfDNA WGS数据准确推断cfDNA甲基化水平和起源组织状态,尤其是对CpG富集区域的预测性能最佳。FinaleMe克服了亚硫酸氢盐转化的障碍,并能够利用大量现有、公开的cfDNA基因组数据集进行表观遗传分析。该研究提供了一种全新的方法,可以对材料有限或只能进行基因组分析的情况下,对cfDNA样本进行甲基化分析。
参考文献:
Liu, Y., Reed, S.C., Lo, C. et al. FinaleMe: Predicting DNA methylation by the fragmentation patterns of plasma cell-free DNA. Nat Commun 15, 2790 (2024). https://doi.org/10.1038/s41467-024-47196-6
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言















#cfDNA# #DNA甲基化#
11